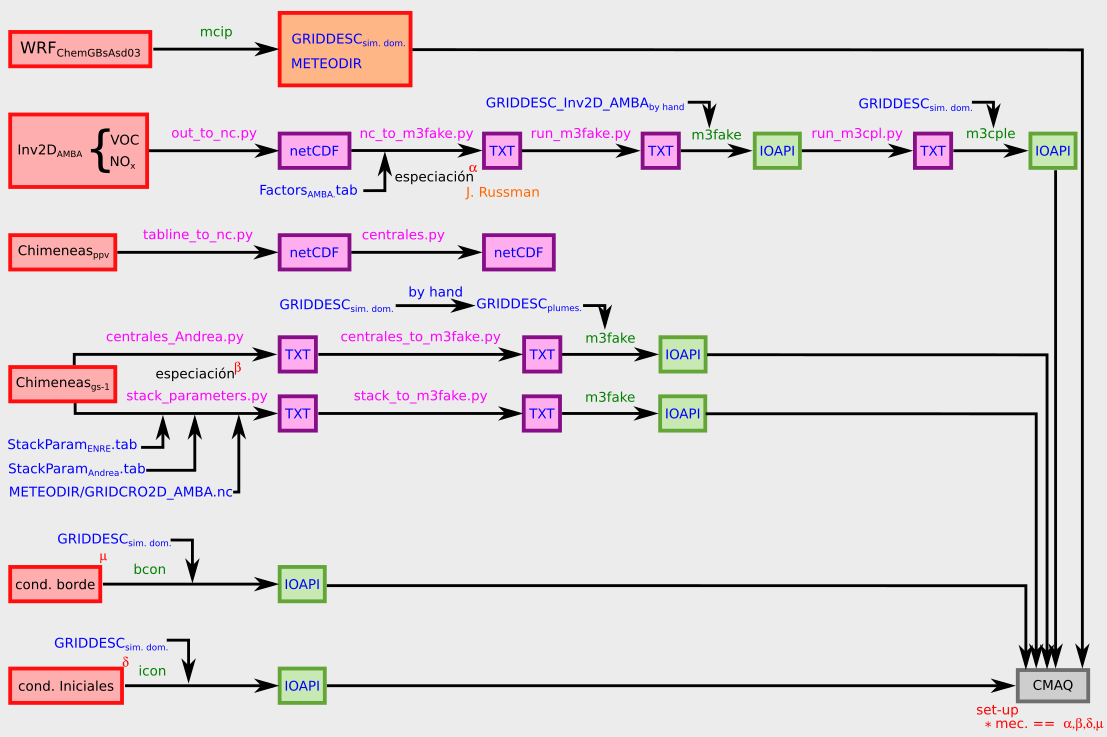
CMAQ/pruebaAMBA
Prueba de simulado en AMBA
WORKDIR = /home/solange.luque/estudios/CMAQ-AMBA/prueba
Contenido |
Datos atmosféricos
Se parte de simulaciones de WRF ya realizadas ver referencia.
Datos en:
/home/solange.luque/salidas/lluis.fita/estudios/ChemGBsAs/sims/weeks/20121110/control
mcip
Se utiliza el programa de CMAQ mcip (MCIP-epa) para generar los archivos con las condiciones atmosféricas necesarias para CMAQ. Seguimos los mismos pasos que [1]
Tener la script para correr el programa:
export CMAQ_HOME=/home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/ cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/PREP/mcip/scripts diff run_mcip.csh run_mcip.csh.old 125,128c125 < # config_cmaq.csh modifies $CMAQ_HOME < set val = $CMAQ_HOME < source $CMAQ_HOME/config_cmaq.csh intel < set CMAQ_HOME = $val --- > source $CMAQ_HOME/config_cmaq.csh 129a127,129 > set APPL = 160702 > set CoordName = LamCon_40N_97W # 16-character maximum > set GridName = 2016_12SE1 # 16-character maximum 131,136c131,132 < set APPL = AMBA < set CoordName = ChemGBsAs_d03 # 16-character maximum < set GridName = ChemGBsAs_d03_cp # 16-character maximum < < set DataPath = /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA < set InMetDir = $DataPath/wrf --- > set DataPath = $CMAQ_DATA > set InMetDir = $DataPath/wrf 138c134 < set OutDir = $DataPath/mcip/$APPL --- > set OutDir = $DataPath/mcip/$GridName 142,143d137 < < 163,165c157,159 < set InMetFiles = ( $InMetDir/wrfout_d03_2012-11-14_12:00:00 \ < $InMetDir/wrfout_d03_2012-11-15_00:00:00 \ < $InMetDir/wrfout_d03_2012-11-15_12:00:00 ) --- > set InMetFiles = ( $InMetDir/subset_wrfout_d01_2016-07-01_00:00:00 \ > $InMetDir/subset_wrfout_d01_2016-07-02_00:00:00 \ > $InMetDir/subset_wrfout_d01_2016-07-03_00:00:00 ) 167,168c161,162 < set IfGeo = "T" < set InGeoFile = $InGeoDir/geo_em.d03.nc --- > set IfGeo = "F" > set InGeoFile = $InGeoDir/geo_em_d01.nc 195,196c189,190 < set MCIP_START = 2012-11-15-00:00:00.0000 # [UTC] < set MCIP_END = 2012-11-15-23:00:00.0000 # [UTC] --- > set MCIP_START = 2016-07-02-00:00:00.0000 # [UTC] > set MCIP_END = 2016-07-03-00:00:00.0000 # [UTC] 222c216 < set BTRIM = 5 --- > set BTRIM = 0 262c256 < set WRF_LC_REF_LAT = -999.0 --- > set WRF_LC_REF_LAT = 40.0
NOTA: El intervalo de salida de mcip tiene que ser el mismo que el del inventario porque sino busca con la frecuencia de mcip los datos en el inventario y no los encuentra. Siento que no debiera ser así, pero no funcionaba de otra forma. Otra cosa: La fecha de inicio y final de mcip también tiene que coincidir con la del inventario. Como el inventario empieza a las 00:00 no queda otra que mcip arranque desde ahí también. Para eso, va a necesitar el time step de las 23 del dia anterior así que si o si hay que agregar el fichero de wrf del dia anterior.
Crear enlaces de los datos atmosféricos (los geo_em.d0[n].nc, ya están ahí):
ln -s /home/solange.luque/salidas/lluis.fita/estudios/ChemGBsAs/sims/weeks/20121110/control/wrfout_d03_* /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/wrf/
Ejecutando:
source /opt/load-libs.sh 1 ./run_mcip.csh >& run_mcip.log
Se obtienen los archivos:
ls /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA GRIDBDY2D_AMBA.nc GRIDDESC LUFRAC_CRO_AMBA.nc METCRO2D_AMBA.nc METDOT3D_AMBA.nc SOI_CRO_AMBA.nc GRIDCRO2D_AMBA.nc GRIDDOT2D_AMBA.nc METBDY3D_AMBA.nc METCRO3D_AMBA.nc namelist.mcip
Este fichero GRIDDESC contiene:
' ' 'ChemGBsAs_d03' 2 -35.600 -33.600 -58.400 -58.400 -34.600 ' ' 'ChemGBsAs_d03_cp' 'ChemGBsAs_d03' -63001.098 -57000.910 1000.000 1000.000 120 114 1 ' '
Inventarios
Inventarios de emisiones y prepararlos para CMAQ.
Seguimos los pasos de [2]
El directorio de trabajo en hydra es:
WORKDIR = /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones
Generación GRIDDESC
Generación de los archivos ASCII de información de las proyecciones (GRIDDESC) en las cuáles se encuentran los distintos datos.
inventario
A partir de los datos ya regrillados en los archivos NOXtot.nc y VOCtot.nc. Se leen directamente los datos de los archivos y se rellena la información. En Inventarios_AMBA.griddesc:
! coords --line: name; type, P-alpha, P-beta, P-gamma, xcent, ycent 'AMBA_emis' 1, 0.0D0, 0.0D0, 0.0D0, 0.0D0, 0.0D0 ' ' ! end coords. grids: name; xorig, yorig, xcell, ycell, ncols, nrows, nthik 'AMBA_emis_cp' 'AMBA_emis' -58.99 -35.05 0.01333 0.01 84 84 1 ' ' ! end grids
dominio
A partir de una salida de WRF y con el programa IOAPI wrfgriddesc se genera directamante el archivo GRIDDESC. NO HACE FALTA. VAMOS A USAR DIRECTAMENTE EL QUE SALE DE MCIP
Variables de entorno
setenv PROMPTFLAG no setenv WRFFILE /home/solange.luque/salidas/lluis.fita/estudios/ChemGBsAs/sims/weeks/20121110/control/wrfout_d03_2012-11-13_00:00:00 setenv OUTDESC /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/ChemGBsAs_d03.griddesc setenv CRDNAME ChemGBsAs_d03 setenv CROGRID ChemGBsAs_d03_cpgrd setenv DOTGRID ChemGBsAs_d03_dotgrd setenv STXGRID ChemGBsAs_d03_xstag setenv STYGRID ChemGBsAs_d03_ystag
Ejecutando:
setenv ROOT_IOAPI /home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64/
${ROOT_IOAPI}/wrfgriddesc >& run_wrfgriddesc.log
El contenido de ChemGBsAs_d03.griddesc
! coords --line: name; type, P-alpha, P-beta, P-gamma, xcent, ycent
'ChemGBsAs_d03'
2 -3.5600000D+01 -3.3600000D+01 -5.8400000D+01 -5.8400000D+01 -3.4600000D+01
' ' ! end coords. grids: name; xorig, yorig, xcell, ycell, ncols, nrows, nthik
'ChemGBsAs_d03_cp'
'ChemGBsAs_d03' -6.9013191E+04 -6.3011015E+04 1.0000000E+03 1.0000000E+03 132 126 1
'ChemGBsAs_d03_do'
'ChemGBsAs_d03' -6.9513191E+04 -6.3511015E+04 1.0000000E+03 1.0000000E+03 133 127 1
'ChemGBsAs_d03_xs'
'ChemGBsAs_d03' -6.9513191E+04 -6.3011015E+04 1.0000000E+03 1.0000000E+03 133 126 1
'ChemGBsAs_d03_ys'
'ChemGBsAs_d03' -6.9013191E+04 -6.3511015E+04 1.0000000E+03 1.0000000E+03 132 127 1
' ' ! end grids
Especiación emisiones
Los archivos con las emisiones de VOC y NOx para AMBA, se tienen en valores totales. Se tienen que especificar (disgregar) para cada tipo de fuente de emisión.
Para especiar el inventario grillado se utiliza la script de python nc_to_m3fake.py del GITcimaCMAQ. Además de las tablas con las características de las múltiples fuentes de AMBA recopiladas en los archivos Estimacion_fi_para_el_AMBA_CMAQ.xlsx (estas tablas de características, así cómo también las fuentes ya regrilladas, son fruto de trabajos anteriores de Andrea Pineda Rojas, Laura E. Vengas Air Quality-Models and Applications, 2011, doi: 10.5772/18767)
python3 nc_to_m3fake.py NOXtot.nc NOx /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/ python3 nc_to_m3fake.py VOCtot.nc VOC /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/
De las scripts se generan los archivos que movemos a la nueva carpeta llamada contaminantes junto con el griddesc del inventario Inventarios_AMBA.griddesc:
ls contaminantes/ ACET_AMBA ALD2_AMBA BENZENE_AMBA ETHA_AMBA ETHY_AMBA Inventarios_AMBA.griddesc ISPD_AMBA NO2_AMBA OLE_AMBA PRPA_AMBA TOL_AMBA XYLMN_AMBA ACROLEIN_AMBA ALDX_AMBA BUTADIENE13_AMBA ETH_AMBA FORM_AMBA IOLE_AMBA KET_AMBA NO_AMBA PAR_AMBA TOLU_AMBA
Para especiar el inventario de las fuentes puntuales se utiliza la script de python centrales_Andrea.py del GITcimaCMAQ. Además de las tablas con los valores de las emisiones de las chimeneas recopiladas en los archivos Chimeneas.xlsx. (Esto no corre en el servidor porque usa pandas).
Esto genera los ficheros (Los movemos a la carpeta contaminantes/puntual):
NO2_puntual NO_puntual
Stack parameters
Para las fuentes puntuales aparte necesitamos un fichero que tenga datos de sus características (altura, diámetro, velocidad de salida,etc).
Para eso hay que correr el script stack_parameters.py del GITcimaCMAQ. Para correr necesita el fichero de salida de MCIP GRIDCRO2D_AMBA.nc que tiene los datos geograficos. Además, necesita las tablas con los valores de las chimeneas que están en los archivos Chimeneas.xlsx y DATOS CHIMENEAS.xlsx. (No corre en el servidor porque usa pandas).
Esto genera los archivos:
ls COL LMAYOR STKDM STKVE IFIP LONGITUDE STKFLW XLOCA ISTACK LPING STKHT YLOCA LATITUDE ROW STKCNT STKTK
Los movemos a la carpeta /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/stack_parameters.
Transformación netCDF a IOAPI
La generación de un archivo en formato IOAPI desde datos en ASCII se realiza con el programa m3fake. Dicho programa requiere la introducción de toda la información de cada fuente de emisión. Para las emisiones grillads, a fin de agilizar este proceso, se ha creado la script de python m3fake/run_m3fake.py (disponible desde GITcimaCMAQ) que lee los distintos archivos de las emisiones (ver paso previo), y genera un archivo ASCII el cual se utiliza para correr el m3fake.
Ejecutando (desde ~/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes):
python3 /home/solange.luque/MODELOS/CMAQ/tools/m3fake/run_m3fake.py /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes /home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64/m3fake
El cuál genera el archivo:
args_m3fake.txt
Se corre el m3fake:
export GRIDDESC=Inventarios_AMBA.griddesc $ROOT_IOAPI/m3fake < args_m3fake.txt
El cuál genera el archivo:
inv_ioapi_AMBA
Para las emisiones puntuales el proceso es similar.
Ejecutando (desde ~/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/puntual):
python3 /home/solange.luque/MODELOS/CMAQ/tools/centrales_to_m3fake.py /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/puntual
El cuál genera el archivo:
puntual_args_m3fake.txt
Antes de correr m3fake necesitamos generar otro griddesc con los datos del dominio, pero donde las columnas sean igual a 1 y las filas igual al número de fuentes puntuales. Creamos el archivo puntual.griddesc.
' ' 'puntual' 2 -35.600 -33.600 -58.400 -58.400 -34.600 ' ' 'puntual_cp' 'puntual' -63001.098 -57000.910 1000.000 1000.000 1 44 1 ' '
Ahora sí, corremos m3fake.
Se corre el m3fake:
export GRIDDESC=puntual.griddesc $ROOT_IOAPI/m3fake < puntual_args_m3fake.txt
Esto genera el archivo
point_AMBA
Para los stack_parameters es análogo.
Ejecutando (desde ~/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/stack_parameters):
python3 /home/solange.luque/MODELOS/CMAQ/tools/stack_to_m3fake.py /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/stack_parameters
El cuál genera el archivo:
stack_args_m3fake.txt
Antes de correr m3fake necesitamos generar otro griddesc con los datos del dominio, pero donde las columnas sean igual a 1 y las filas igual al número de fuentes puntuales. Creamos el archivo stack.griddesc.
! coords --line: name; type, P-alpha, P-beta, P-gamma, xcent, ycent
'ChemGBsAs_d03'
2 -3.5600000D+01 -3.3600000D+01 -5.8400000D+01 -5.8400000D+01 -3.4600000D+01
' ' ! end coords. grids: name; xorig, yorig, xcell, ycell, ncols, nrows, nthik
'ChemGBsAs_d03_cp'
'ChemGBsAs_d03' -6.9013191E+04 -6.3011015E+04 1.0000000E+03 1.0000000E+03 1 44 1
'ChemGBsAs_d03_do'
'ChemGBsAs_d03' -6.9513191E+04 -6.3511015E+04 1.0000000E+03 1.0000000E+03 133 127 1
'ChemGBsAs_d03_xs'
'ChemGBsAs_d03' -6.9513191E+04 -6.3011015E+04 1.0000000E+03 1.0000000E+03 133 126 1
'ChemGBsAs_d03_ys'
'ChemGBsAs_d03' -6.9013191E+04 -6.3511015E+04 1.0000000E+03 1.0000000E+03 132 127 1
' ' ! end grids
Ahora sí, corremos m3fake.
Se corre el m3fake:
export GRIDDESC=stack.griddesc $ROOT_IOAPI/m3fake < stack_args_m3fake.txt
Esto genera el archivo
stack_AMBA
Interpolación a dominio de simulación
Para interpolar el inventario al dominio de simulación vamos a trabajar en:
WORKDIR = /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/interpolacion
Necesitamos acá el griddesc del dominio de simulación. Lo podemos linkear:
ln -s /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA mcip.griddesc
Necesitamos también el inventario en IOAPI que acabamos de crear:
ln -s /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/inv_ioapi_AMBA inv_1k
Variables de entorno:
setenv GRIDDESC mcip.griddesc setenv SCALEFAC 1.0
Ahora queda correr el script de python que genera los argumentos para correr m3cple (Si quisieramos cambiarle el nombre a los ficheros hay que modificarlo ahí. En el script o a mano en el .txt, es lo mismo)
python3 /home/solange.luque/MODELOS/CMAQ/tools/run_m3cple.py serv
Esto genera el archivo ASCII
args_m3cple.txt
Ahora sí, se corre finalmente m3cple.
$ROOT_IOAPI/m3cple < args_m3cple.txt
Esto genera el fichero IOAPI:
gridr_inv_1k
C.I. y C.C. químicas
Condiciones de contorno e iniciales para la química atmosférica
El directorio de trabajo en hydra es:
WORKDIR = /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/icbc
Como un primer acercamiento, vamos a usar de condiciones de contorno e iniciales los perfiles verticales que vienen por default con CMAQ. Los linkeamos acá:
ln -s /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/PREP/bcon/src/profile/avprofile_* .
Deberíamos tener:
[1] avprofile_cb6r3m_ae7_kmtbr_hemi2016_v53beta2_m3dry_col051_row068.csv [2] avprofile_saprc07tc_ae6_aq_derived_from_cb6r3m_ae7_kmtbr_hemi2016_v53beta2_m3dry_col051_row068.csv [3] avprofile_racm_ae6_aq_derived_from_cb6r3m_ae7_kmtbr_hemi2016_v53beta2_m3dry_col051_row068.csv [4] avprofile_saprc07tic_ae7i_aq_derived_from_cb6r3m_ae7_kmtbr_hemi2016_v53beta2_m3dry_col051_row068.csv
Cada uno de estos está calculado usando diferentes mecanismos. Debiéramos elegir uno, que tiene que coincidir con la especiación / mecanismo de las emisiones propias. Mirar figuras (número coincide con nombre de archivo)
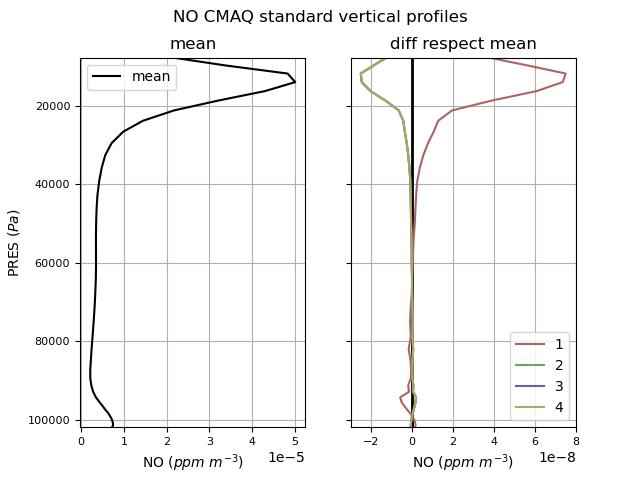 |
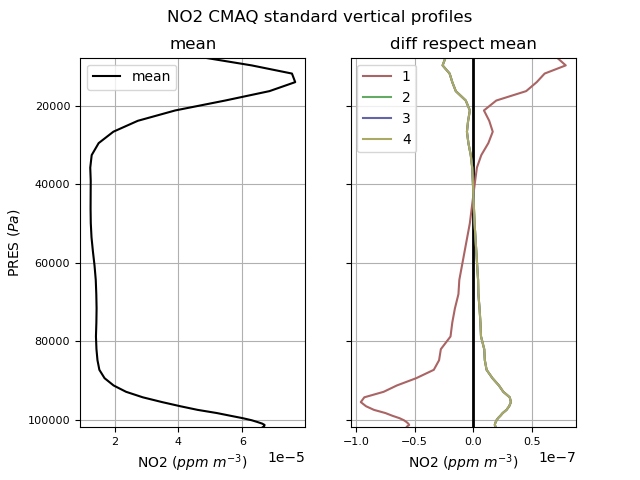
|
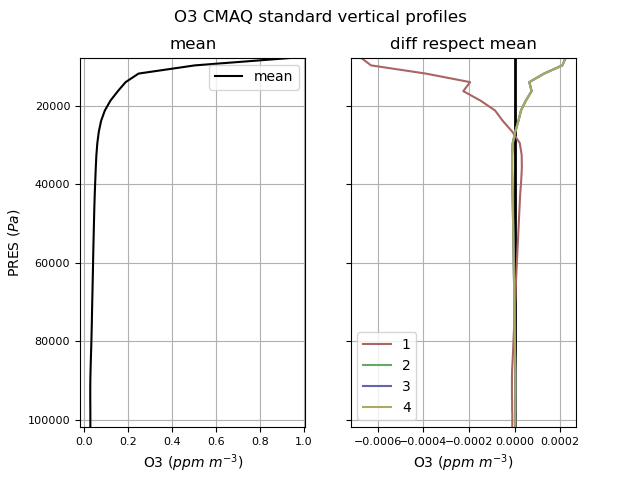 |
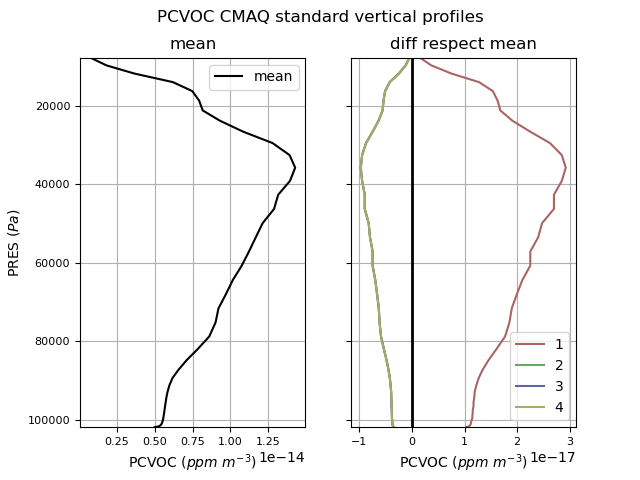
|
Se tiene que transformar formato IOAPI utilizando las herramientas icon (como en [3]) y bcon (como en [4])
Empecemos por las condiciones iniciales. La carpeta de trabajo es:
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/PREP/icon/scripts
Hay que modificar el script run_icon.csh. Para eso copiamos la versión vieja y modificamos. Si queremos cambiar el perfil que usamos (para usar otro mecanismo), es acá también. Por ahora lo dejé así con cb6r3m_ae7.
cp run_icon.csh run_icon.csh.old
diff run_icon.csh run_icon.csh.old
< set APPL = AMBA #> Application Name
< set ICTYPE = profile #> Initial conditions type [profile|regrid]
---
> set APPL = 2016_12SE1 #> Application Name
> set ICTYPE = regrid #> Initial conditions type [profile|regrid]
41c41
< setenv GRID_NAME ChemGBsAs_d03_cp #> check GRIDDESC file for GRID_NAME options
---
> setenv GRID_NAME SE53BENCH #> check GRIDDESC file for GRID_NAME options
43c43
< setenv GRIDDESC /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA/GRIDDESC
---
> setenv GRIDDESC /work/MOD3DATA/SE53BENCH/met/mcip/GRIDDESC
88c88
< set DATE = "2012-11-15"
---
> set DATE = "2016-07-01"
104c104
< setenv MET_CRO_3D_FIN /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA/METCRO3D_AMBA.nc
---
> setenv MET_CRO_3D_FIN /work/MOD3DATA/SE53BENCH/met/mcip/METCRO3D_${YYMMDD}.nc
Y ahora ejecutamos:
./run_icon.csh |& tee run_icon.log
Esto genera el fichero:
/home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/icon/ICON_v54_AMBA_profile_20121115
Ahora a las condiciones de borde. Hacemos lo mismo: Nos vamos a la carpeta de bcon y copiamos el run_bcon.csh.
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/PREP/bcon/scripts cp run_bcon.csh run_bcon.csh.old
Ahora lo modificamos. Acá también si queremos cambiar el perfil que usamos (para usar otro mecanismo), hay que cambiarlo. Por ahora lo dejé así con cb6r3m_ae7.
diff run_bcon.csh run_bcon.csh.old
32,33c32,33
< set APPL = AMBA #> Application Name
< set BCTYPE = profile #> Boundary condition type [profile|regrid]
---
> set APPL = 2016_12SE1 #> Application Name
> set BCTYPE = regrid #> Boundary condition type [profile|regrid]
41c41
< setenv GRID_NAME ChemGBsAs_d03_cp #> check GRIDDESC file for GRID_NAME options
---
> setenv GRID_NAME SE53BENCH #> check GRIDDESC file for GRID_NAME options
43c43
< setenv GRIDDESC /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA/GRIDDESC
---
> setenv GRIDDESC /work/MOD3DATA/SE53BENCH/met/mcip/GRIDDESC
88c88
< set DATE = "2012-11-15"
---
> set DATE = "2016-07-01"
105c105
< setenv MET_BDY_3D_FIN /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/mcip/AMBA/METBDY3D_AMBA.nc
---
> setenv MET_BDY_3D_FIN /work/MOD3DATA/SE53BENCH/met/mcip/METBDY3D_${YYMMDD}.nc
Ahora ejecutamos.
./run_bcon.csh |& tee run_bcon.log
Esto crea el fichero:
/home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/bcon/BCON_v54_AMBA_profile_20121115
Ahora copiamos estos dos ficheros en el directorio de trabajo
cp /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/bcon/BCON_v54_AMBA_profile_20121115 /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/icbc/BCON_v54_AMBA_profile_20121115 cp /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/icon/ICON_v54_AMBA_profile_20121115 /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/icbc/ICON_v54_AMBA_profile_20121115
Configuración CMAQ
Configurar CMAQ para determinar opciones de química y física.
Primero creamos la carpeta AMBA:
mkdir /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/AMBA
En esa carpeta vamos a linkear los inputs.
ln -s ~/estudios/CMAQ-AMBA/prueba/DATA/icbc icbc ln -s ~/estudios/CMAQ-AMBA/prueba/DATA/mcip mcip ln -s ~/estudios/CMAQ-AMBA/prueba/DATA/wrf wrf ln -s ~/estudios/CMAQ-AMBA/prueba/DATA/emisiones/ChemGBsAs_d03.griddesc GRIDDESC
Creamos una carpeta para las emisiones grilladas:
mkdir emis cd emis mkdir gridded_area cd gridded_area mkdir gridded
Linkeamos el inventario ahí:
ln -s /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/interpolacion/gridr_inv_1k /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/AMBA/emis/gridded_area/gridded/gridded_AMBA
Ahora creamos una carpeta para las emisiones puntuales.
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/AMBA/emis mkdir inln_point cd inln_point mkdir pt_oilgas #no sé si esta la categoría que quiero, pero por ahora... cd pt_oilgas
Linkeo ahí el inventario.
ln -s /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/puntual/point_AMBA /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/AMBA/emis/inln_point/pt_oilgas/point_AMBA
Ahora generamos una carpeta para los stack parameters.
cd .. mkdir stack_groups
Linkeamos ahí los stack_parameters.
ln -s /home/solange.luque/estudios/CMAQ-AMBA/prueba/DATA/emisiones/contaminantes/stack_parameters/stack_AMBA /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/AMBA/emis/inln_point/stack_groups/stack_AMBA
Primero hay que editar bldit_cctm.csh con las opciones que querramos. Esto está acá:
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts cp bldit_cctm.csh bldit_cctm.csh.old diff bldit_cctm.csh bldit_cctm.csh.old 134c134 < setenv Mechanism cb6r3_ae7_aq #> chemical mechanism (see $CMAQ_MODEL/CCTM/src/MECHS) --- > setenv Mechanism cb6r5_ae7_aq #> chemical mechanism (see $CMAQ_MODEL/CCTM/src/MECHS)
Lo corremos:
./bldit_cctm.csh intel >& bldit_cctm_AMBA.log
Nota: Si es una recompilación, hay que borrar todo antes.
rm -rf BLD_CCTM_v54_intel/*
Esto debiera crear al ejecutable:
/home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/CCTM_v54.exe
Ahora hay que editar los paths de los inputs. Esto está definido en el .csh que se corre con el modelo. Para eso nos copiamos primero uno de referencia y lo vamos a modificar. Vamos a tener un script donde solo tenemos el inventario de la grilla (run_AMBA_grilla.csh) y otro donde tenemos tanto la grilla como las fuentes puntuales (run_AMBA_grilla.csh). Para solo la grilla:
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts
cp run_cctm_Bench_2016_12SE1.csh run_AMBA_grilla.csh
diff run_cctm_Bench_2016_12SE1.csh run_AMBA.csh
38,39c38,39
< set MECH = cb6r5_ae7_aq #> Mechanism ID
< set APPL = Bench_2016_12SE1 #> Application Name (e.g. Gridname)
---
> set MECH = cb6r3_ae7_aq #> Mechanism ID
> set APPL = AMBA #> Application Name (e.g. Gridname)
57c57
< setenv INPDIR ${CMAQ_DATA}/2016_12SE1 #> Input Directory
---
> setenv INPDIR ${CMAQ_DATA}/AMBA #> Input Directory
75,76c75,76
< set START_DATE = "2016-07-01" #> beginning date (July 1, 2016)
< set END_DATE = "2016-07-01" #> ending date (July 1, 2016)
---
> set START_DATE = "2012-11-15" #> beginning date (July 1, 2016)
> set END_DATE = "2012-11-15" #> ending date (July 1, 2016)
121c121
< setenv GRID_NAME 2016_12SE1 #> check GRIDDESC file for GRID_NAME options
---
> setenv GRID_NAME ChemGBsAs_d03_cp #> check GRIDDESC file for GRID_NAME options
150c150
< setenv CTM_OCEAN_CHEM Y #> Flag for ocean halgoen chemistry and sea spray aerosol emissions [ default: Y ]
---
> setenv CTM_OCEAN_CHEM N #> Flag for ocean halgoen chemistry and sea spray aerosol emissions [ default: Y ]
158c158
< setenv CTM_ABFLUX Y #> ammonia bi-directional flux for in-line deposition
---
> setenv CTM_ABFLUX N #> ammonia bi-directional flux for in-line deposition
169c169
< setenv CTM_BIOGEMIS_BE Y #> calculate in-line biogenic emissions with BEIS [ default: N ]
---
> setenv CTM_BIOGEMIS_BE N #> calculate in-line biogenic emissions with BEIS [ default: N ]
233c233
< set METpath = $INPDIR/met/mcipv5.0 #> meteorology input directory
---
> set METpath = $INPDIR/mcip/AMBA #> meteorology input directory
283c283
< setenv ICFILE ICON_20160630_bench.nc
---
> setenv ICFILE ICON_v54_AMBA_profile_20121115
292c292
< set BCFILE = BCON_${YYYYMMDD}_bench.nc
---
> set BCFILE = BCON_v54_AMBA_profile_20121115
304,312c304,312
< setenv GRID_BDY_2D $METpath/GRIDBDY2D_${YYMMDD}.nc # GRID files are static, not day-specific
< setenv GRID_CRO_2D $METpath/GRIDCRO2D_${YYMMDD}.nc
< setenv GRID_CRO_3D $METpath/GRIDCRO3D_${YYMMDD}.nc
< setenv GRID_DOT_2D $METpath/GRIDDOT2D_${YYMMDD}.nc
< setenv MET_CRO_2D $METpath/METCRO2D_${YYMMDD}.nc
< setenv MET_CRO_3D $METpath/METCRO3D_${YYMMDD}.nc
< setenv MET_DOT_3D $METpath/METDOT3D_${YYMMDD}.nc
< setenv MET_BDY_3D $METpath/METBDY3D_${YYMMDD}.nc
< setenv LUFRAC_CRO $METpath/LUFRAC_CRO_${YYMMDD}.nc
---
> setenv GRID_BDY_2D $METpath/GRIDBDY2D_AMBA.nc # GRID files are static, not day-specific
> setenv GRID_CRO_2D $METpath/GRIDCRO2D_AMBA.nc
> setenv GRID_CRO_3D $METpath/GRIDCRO3D_AMBA.nc
> setenv GRID_DOT_2D $METpath/GRIDDOT2D_AMBA.nc
> setenv MET_CRO_2D $METpath/METCRO2D_AMBA.nc
> setenv MET_CRO_3D $METpath/METCRO3D_AMBA.nc
> setenv MET_DOT_3D $METpath/METDOT3D_AMBA.nc
> setenv MET_BDY_3D $METpath/METBDY3D_AMBA.nc
> setenv LUFRAC_CRO $METpath/LUFRAC_CRO_AMBA.nc
344c344
< setenv CMAQ_MASKS $SZpath/OCEAN_${MM}_L3m_MC_CHL_chlor_a_SE53BENCH.nc #> horizontal grid-dependent ocean file
---
> #setenv CMAQ_MASKS $SZpath/OCEAN_${MM}_L3m_MC_CHL_chlor_a_SE53BENCH.nc #> horizontal grid-dependent ocean file
347,348c347,348
< setenv N_EMIS_GR 2
< set EMISfile = emis_mole_all_${YYYYMMDD}_cb6_bench.nc
---
> setenv N_EMIS_GR 1
> set EMISfile = gridded_AMBA
351c351
< setenv GR_EM_SYM_DATE_001 F # To change default behaviour please see Users Guide for EMIS_SYM_DATE
---
> setenv GR_EM_SYM_DATE_001 Y # To change default behaviour please see Users Guide for EMIS_SYM_DATE
353,356c353,356
< set EMISfile = emis_mole_rwc_${YYYYMMDD}_12US1_cmaq_cb6_2016ff_16j.nc
< setenv GR_EMIS_002 ${EMISpath2}/${EMISfile}
< setenv GR_EMIS_LAB_002 GR_RES_FIRES
< setenv GR_EM_SYM_DATE_002 F # To change default behaviour please see Users Guide for EMIS_SYM_DATE
---
> #set EMISfile = emis_mole_rwc_${YYYYMMDD}_12US1_cmaq_cb6_2016ff_16j.nc
> #setenv GR_EMIS_002 ${EMISpath2}/${EMISfile}
> #setenv GR_EMIS_LAB_002 GR_RES_FIRES
> #setenv GR_EM_SYM_DATE_002 F # To change default behaviour please see Users Guide for EMIS_SYM_DATE
359c359
< setenv N_EMIS_PT 8 #> Number of elevated source groups
---
> #setenv N_EMIS_PT 8 #> Number of elevated source groups
361,362c361,362
< set STKCASEG = 12US1_2016ff_16j # Stack Group Version Label
< set STKCASEE = 12US1_cmaq_cb6_2016ff_16j # Stack Emission Version Label
---
> #set STKCASEG = 12US1_2016ff_16j # Stack Group Version Label
> #set STKCASEE = 12US1_cmaq_cb6_2016ff_16j # Stack Emission Version Label
365,372c365,372
< setenv STK_GRPS_001 $IN_PTpath/stack_groups/stack_groups_ptnonipm_${STKCASEG}.nc
< setenv STK_GRPS_002 $IN_PTpath/stack_groups/stack_groups_ptegu_${STKCASEG}.nc
< setenv STK_GRPS_003 $IN_PTpath/stack_groups/stack_groups_othpt_${STKCASEG}.nc
< setenv STK_GRPS_004 $IN_PTpath/stack_groups/stack_groups_ptagfire_${YYYYMMDD}_${STKCASEG}.nc
< setenv STK_GRPS_005 $IN_PTpath/stack_groups/stack_groups_ptfire_${YYYYMMDD}_${STKCASEG}.nc
< setenv STK_GRPS_006 $IN_PTpath/stack_groups/stack_groups_ptfire_othna_${YYYYMMDD}_${STKCASEG}.nc
< setenv STK_GRPS_007 $IN_PTpath/stack_groups/stack_groups_pt_oilgas_${STKCASEG}.nc
< setenv STK_GRPS_008 $IN_PTpath/stack_groups/stack_groups_cmv_c3_${STKCASEG}.nc
---
> #setenv STK_GRPS_001 $IN_PTpath/stack_groups/stack_groups_ptnonipm_${STKCASEG}.nc
> #setenv STK_GRPS_002 $IN_PTpath/stack_groups/stack_groups_ptegu_${STKCASEG}.nc
> #setenv STK_GRPS_003 $IN_PTpath/stack_groups/stack_groups_othpt_${STKCASEG}.nc
> #setenv STK_GRPS_004 $IN_PTpath/stack_groups/stack_groups_ptagfire_${YYYYMMDD}_${STKCASEG}.nc
> #setenv STK_GRPS_005 $IN_PTpath/stack_groups/stack_groups_ptfire_${YYYYMMDD}_${STKCASEG}.nc
> #setenv STK_GRPS_006 $IN_PTpath/stack_groups/stack_groups_ptfire_othna_${YYYYMMDD}_${STKCASEG}.nc
> #setenv STK_GRPS_007 $IN_PTpath/stack_groups/stack_groups_pt_oilgas_${STKCASEG}.nc
> #setenv STK_GRPS_008 $IN_PTpath/stack_groups/stack_groups_cmv_c3_${STKCASEG}.nc
375,382c375,382
< setenv STK_EMIS_001 $IN_PTpath/ptnonipm/inln_mole_ptnonipm_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_002 $IN_PTpath/ptegu/inln_mole_ptegu_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_003 $IN_PTpath/othpt/inln_mole_othpt_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_004 $IN_PTpath/ptagfire/inln_mole_ptagfire_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_005 $IN_PTpath/ptfire/inln_mole_ptfire_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_006 $IN_PTpath/ptfire_othna/inln_mole_ptfire_othna_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_007 $IN_PTpath/pt_oilgas/inln_mole_pt_oilgas_${YYYYMMDD}_${STKCASEE}.nc
< setenv STK_EMIS_008 $IN_PTpath/cmv_c3/inln_mole_cmv_c3_${YYYYMMDD}_${STKCASEE}.nc
---
> #setenv STK_EMIS_001 $IN_PTpath/ptnonipm/inln_mole_ptnonipm_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_002 $IN_PTpath/ptegu/inln_mole_ptegu_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_003 $IN_PTpath/othpt/inln_mole_othpt_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_004 $IN_PTpath/ptagfire/inln_mole_ptagfire_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_005 $IN_PTpath/ptfire/inln_mole_ptfire_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_006 $IN_PTpath/ptfire_othna/inln_mole_ptfire_othna_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_007 $IN_PTpath/pt_oilgas/inln_mole_pt_oilgas_${YYYYMMDD}_${STKCASEE}.nc
> #setenv STK_EMIS_008 $IN_PTpath/cmv_c3/inln_mole_cmv_c3_${YYYYMMDD}_${STKCASEE}.nc
385,392c385,392
< setenv STK_EMIS_LAB_001 PT_NONEGU
< setenv STK_EMIS_LAB_002 PT_EGU
< setenv STK_EMIS_LAB_003 PT_OTHER
< setenv STK_EMIS_LAB_004 PT_AGFIRES
< setenv STK_EMIS_LAB_005 PT_FIRES
< setenv STK_EMIS_LAB_006 PT_OTHFIRES
< setenv STK_EMIS_LAB_007 PT_OILGAS
< setenv STK_EMIS_LAB_008 PT_CMV
---
> #setenv STK_EMIS_LAB_001 PT_NONEGU
> #setenv STK_EMIS_LAB_002 PT_EGU
> #setenv STK_EMIS_LAB_003 PT_OTHER
> #setenv STK_EMIS_LAB_004 PT_AGFIRES
> #setenv STK_EMIS_LAB_005 PT_FIRES
> #setenv STK_EMIS_LAB_006 PT_OTHFIRES
> #setenv STK_EMIS_LAB_007 PT_OILGAS
> #setenv STK_EMIS_LAB_008 PT_CMV
397,404c397,404
< setenv STK_EM_SYM_DATE_001 T
< setenv STK_EM_SYM_DATE_002 T
< setenv STK_EM_SYM_DATE_003 T
< setenv STK_EM_SYM_DATE_004 T
< setenv STK_EM_SYM_DATE_005 T
< setenv STK_EM_SYM_DATE_006 T
< setenv STK_EM_SYM_DATE_007 T
< setenv STK_EM_SYM_DATE_008 T
---
> #setenv STK_EM_SYM_DATE_001 T
> #setenv STK_EM_SYM_DATE_002 T
> #setenv STK_EM_SYM_DATE_003 T
> #setenv STK_EM_SYM_DATE_004 T
> #setenv STK_EM_SYM_DATE_005 T
> #setenv STK_EM_SYM_DATE_006 T
> #setenv STK_EM_SYM_DATE_007 T
> #setenv STK_EM_SYM_DATE_008 T
444c444
< setenv OCEAN_1 $SZpath/OCEAN_${MM}_L3m_MC_CHL_chlor_a_SE53BENCH.nc #> horizontal grid-dependent ocean file
---
> #setenv OCEAN_1 $SZpath/OCEAN_${MM}_L3m_MC_CHL_chlor_a_SE53BENCH.nc #> horizontal grid-dependent ocean file
710c710,711
< ( /usr/bin/time -p mpirun -np $NPROCS $BLD/$EXEC ) |& tee buff_${EXECUTION_ID}.txt
---
> # ( /usr/bin/time -p mpirun -np $NPROCS $BLD/$EXEC ) |& tee buff_${EXECUTION_ID}.txt
> (mpirun -np $NPROCS $BLD/$EXEC ) |& tee buff_${EXECUTION_ID}.txt
Para el script con tanto la grilla como las fuentes puntuales:
cp run_AMBA_grilla.csh run_AMBA.csh
diff run_AMBA_grilla.csh run_AMBA.csh
359c359
< #setenv N_EMIS_PT 8 #> Number of elevated source groups
---
> setenv N_EMIS_PT 1 #> Number of elevated source groups
361,362c361,362
< #set STKCASEG = 12US1_2016ff_16j # Stack Group Version Label
< #set STKCASEE = 12US1_cmaq_cb6_2016ff_16j # Stack Emission Version Label
---
> set STKCASEG = AMBA # Stack Group Version Label
> set STKCASEE = AMBA # Stack Emission Version Label
365c365
< #setenv STK_GRPS_001 $IN_PTpath/stack_groups/stack_groups_ptnonipm_${STKCASEG}.nc
---
> setenv STK_GRPS_001 $IN_PTpath/stack_groups/stack_AMBA
381c381
< #setenv STK_EMIS_007 $IN_PTpath/pt_oilgas/inln_mole_pt_oilgas_${YYYYMMDD}_${STKCASEE}.nc
---
> setenv STK_EMIS_001 $IN_PTpath/pt_oilgas/point_AMBA
391c391
< #setenv STK_EMIS_LAB_007 PT_OILGAS
---
> setenv STK_EMIS_LAB_001 PT_OILGAS
397c397
< #setenv STK_EM_SYM_DATE_001 T
---
> setenv STK_EM_SYM_DATE_001 T
NOTAS sobre la configuración:
- Acerca de la estabilidad numérica y CFLs, leer este post en el foro CMAS
Escabilidad en el servidor
Si queremos cambiar el número de procesos que queremos usar para correr el modelo esto también se puede cambiar en run_AMBA.csh, cambiando los valores de:
@ NPCOL = 8; @ NPROW = 4
Donde el número de procesos totales será NPROCS = NPCOL*NPROW.
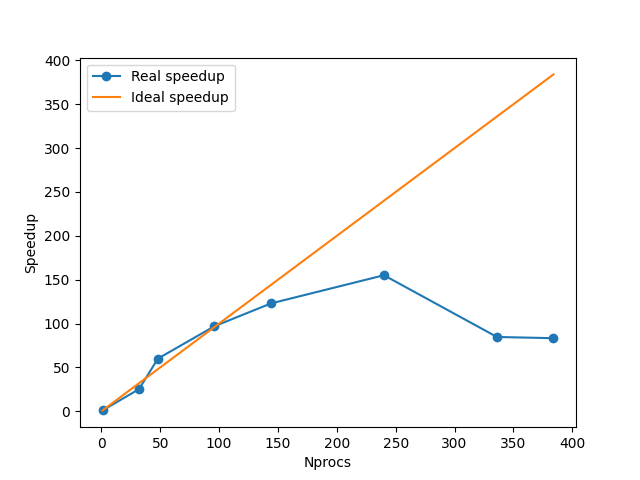
Para entender cuál es el número más apropiado de procesadores se corrió el dia de prueba varias veces con distintos valores de NPROCS. Luego, se calculó el speedup (S) para cada caso donde S = T(1)/T(n) siendo T(1) el tiempo que tarda en correr con un solo procesador y T(n) el tiempo que tarda con n. Para conocer T(1), se corrió el modelo con un solo procesador por unas pocas horas y luego se promedió el tiempo que tardaba por paso para saber cuanto tardaría aproximadamente en terminar de correr el dia entero.
Idealmente, el speedup crece de forma lineal con el número de procesadores.
Como se puede ver, en el rango aproximado de NPROCS = 50-100, el speed up real es levemente mayor que el ideal. Esto no debiera pasar, pero se debe muy probablemente a que el valor de T(1) utilizado para hacer los cálculos es aproximado y no real. Por el otro lado, a partir de NPROCS = 150, el speedup real se empieza a alejar cada vez más del ideal. Sin embargo, hasta NPROCS = 244 se sigue viendo mejora en el tiempo de corrida. Por encima de este valor, el modelo tarda más en correr.
Simulado CMAQ
Ahora sí, solo queda correr el modelo. El directorio de trabajo es
WORKDIR = /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts
Para correr en la cola de Hydra necesitamos un script. Nos copiamos uno que ya hay:
cp launch_cctm_intel.pbs launch_cctm_intel_AMBA.pbs
Lo modificamos:
diff launch_cctm_intel.pbs launch_cctm_intel_AMBA.pbs 38c38 < ./run_cctm_Bench_2018_12NE3.csh >& run_Bench_2018-12NE3.log --- > ./run_AMBA.csh >& run_AMBA.log
Si queremos cambiar el número de procesos también hay que cambiar las líneas:
#PBS nodes=1:ppn=32
Donde se debe cumplir que nodes*ppn = NPROCS = NPCOL*NPROW
Hay un par de tablas que necesitamos del mecanismo que estamos usando En este caso cb6r3_ae7_aq). Se pueden encontrar acá,
Las creamos y le copiamos los datos:
nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/CSQY_DATA_cb6r3_ae7_aq nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/GC_cb6r3_ae7_aq.nml nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/AE_cb6r3_ae7_aq.nml nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/NR_cb6r3_ae7_aq.nml nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/CMAQ_Control_DESID_cb6r3_ae7_aq.nml nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/SpecDef_Dep_cb6r3_ae7_aq.txt nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/SpecDef_cb6r3_ae7_aq.txt nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/mech_cb6r3_ae7_aq.def nano /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel/pa_cb6r3_ae7_aq.ctl
Ahora sí, corremos:
qsub launch_cctm_intel_AMBA.pbs
Si hay algún error, conviene mirar en el log de que nodo está el problema:
grep -i error CTM_LOG*
Si todo salió bien, se deberían generar los ficheros en el directorio:
/home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/data/output_CCTM_v54_intel_AMBA
Deberíamos tener los siguientes ficheros:
CCTM_ACONC_v54_intel_AMBA_20121115.nc CCTM_AELMO_v54_intel_AMBA_20121115.nc CCTM_BUDGET_v54_intel_AMBA_20121115.txt CCTM_CONC_v54_intel_AMBA_20121115.nc CCTM_DRYDEP_v54_intel_AMBA_20121115.nc CCTM_v54_intel_AMBA_20121115.cfg CCTM_WETDEP1_v54_intel_AMBA_20121115.nc LOGS
La data en estos ficheros eso:
ACONC: Hourly average concentration file AELMO: Average hourly ELMO (Explicit and Lumped Model Output) output file. Este fichero es opcional BUDGET: Budget Tool Output File. Outputs domain-wide changes for user-specified species every output time step. Opcional CONC: Hourly instantaneous concentration file DRYDEP: Hourly cumulative dry deposition file. Opcional WETDEP: Hourly cumulative wet deposition file. Opcional