CMAQInvCbCi
(→Megan en sí) |
(→Acoplado con CMAQ) |
||
| (No se muestran 48 ediciones intermedias realizadas por un usuario) | |||
| Línea 1.135: | Línea 1.135: | ||
===Megan 3.2=== |
===Megan 3.2=== |
||
| − | Megan 3.2 [[https://bai.ess.uci.edu/megan/data-and-code/megan32]] tiene unos pre procesadores. En total son 3 códigos a bajar: Un pre procesador de Fortran (Que agarra los datasets y los mapea al dominio de simulación), el pre procesador de Python (Que Calcula los factores de emisión) y Megan propiamente dicho. |
+ | Megan 3.2 [[https://bai.ess.uci.edu/megan/data-and-code/megan32]] tiene dos pre procesadores. En total son 3 códigos a bajar: Un pre procesador de Fortran (Que agarra los datasets y los mapea al dominio de simulación), el pre procesador de Python (Que Calcula los factores de emisión) y Megan propiamente dicho. |
| − | ==== Compilación pre procesador de Fortran ==== |
+ | ==== Pre procesador de Fortran ==== |
| + | |||
| + | ===== Compilación ===== |
||
Se descarga de acá [[https://drive.google.com/file/d/1R0oPCqzj8eNhg3ltfy9LRrGcyA4kbifF/view]] y acá [[https://drive.google.com/file/d/1ONtMjwxr9N7bnNDYaaPs1JEt19pvN65P/view]] |
Se descarga de acá [[https://drive.google.com/file/d/1R0oPCqzj8eNhg3ltfy9LRrGcyA4kbifF/view]] y acá [[https://drive.google.com/file/d/1ONtMjwxr9N7bnNDYaaPs1JEt19pvN65P/view]] |
||
| Línea 1.201: | Línea 1.201: | ||
<pre style = "shell"> |
<pre style = "shell"> |
||
| − | diff prepmegan4cmaq_cantype.f90 prepmegan4cmaq_cantype.f90.old |
+ | diff prepmegan4cmaq_cantype.f90 prepmegan4cmaq_cantype.f90.old |
| − | 979c979 |
+ | 190,191c190,191 |
| − | < write (99, '(3(I9,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, & |
+ | < missing_value = -128 |
| + | < !read(treefillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > !missing_value = -1. |
||
| + | > read(treefillvalue,"(f7.0)") missing_value |
||
| + | 195,196c195 |
||
| + | < missing_value = 0. |
||
| + | < !read(NTfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(NTfillvalue,"(f7.0)") missing_value |
||
| + | 202,203c201 |
||
| + | < missing_value = -1 |
||
| + | < !read(TRTfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(TRTfillvalue,"(f7.0)") missing_value |
||
| + | 206d203 |
||
| + | < missing_value = -128 |
||
| + | 209c206 |
||
| + | < !read(shrubfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(shrubfillvalue,"(f7.0)") missing_value |
||
| + | 213c210 |
||
| + | < !read(cropfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(cropfillvalue,"(f7.0)") missing_value |
||
| + | 217c214 |
||
| + | < !read(grassfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(grassfillvalue,"(f7.0)") missing_value |
||
| + | 982c979 |
||
| + | < write (99, '(3(I0,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, & |
||
--- |
--- |
||
> write (99, '(3(I,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, & |
> write (99, '(3(I,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, & |
||
</pre> |
</pre> |
||
| + | Ojo, acá también le estoy harcodeando los missing_values para cada archivo. En caso de cambiar, hay que cambiarlos. |
||
Y en <code>prepmegan4cmaq_w126.f90</code>. |
Y en <code>prepmegan4cmaq_w126.f90</code>. |
||
| Línea 1.218: | Línea 1.219: | ||
> write (99, '(3(I,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, & |
> write (99, '(3(I,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, & |
||
</pre> |
</pre> |
||
| + | |||
| + | Y en <code> prepmegan4cmaq_grwform.f90</code> |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | diff prepmegan4cmaq_grwform.f90 prepmegan4cmaq_grwform.f90.old |
||
| + | 210,211c210,211 |
||
| + | < missing_value = -128 |
||
| + | < !read(treefillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > !missing_value = -1. |
||
| + | > read(treefillvalue,"(f7.0)") missing_value |
||
| + | 215c215 |
||
| + | < !read(shrubfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(shrubfillvalue,"(f7.0)") missing_value |
||
| + | 219c219 |
||
| + | < !read(cropfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(cropfillvalue,"(f7.0)") missing_value |
||
| + | 223c223 |
||
| + | < !read(grassfillvalue,"(f7.0)") missing_value |
||
| + | --- |
||
| + | > read(grassfillvalue,"(f7.0)") missing_value |
||
| + | |||
| + | </pre> |
||
| + | |||
| + | Ojo con este: Le setee -128 como missing_value porque es el que tienen los ficheros que voy a usar (todos el mismo). Si no fuera el caso, habría que leerlo de los ficheros como estaba antes, pero por algún motivo no funcionaba. |
||
Y ahora corremos: |
Y ahora corremos: |
||
| Línea 1.286: | Línea 1.314: | ||
./make_util prepmegan4cmaq_landtype.x |
./make_util prepmegan4cmaq_landtype.x |
||
./make_util prepmegan4cmaq_nitrogen.x |
./make_util prepmegan4cmaq_nitrogen.x |
||
| + | </pre> |
||
| + | |||
| + | ===== Inputs ===== |
||
| + | |||
| + | El pre procesador de Fortran necesita varios inputs: |
||
| + | |||
| + | 1) Leaf Index Area [[https://bai.ess.uci.edu/megan/data-and-code/lai]]. Hay una versión global, pero solo sirve para Megan2. Tiene otro formato (no está en netcdf). En cambio, lo que se puede hacer es generarse los netcdfs necesarios con datos de MODIS. De todas formas, no son obligatorios para usar esta versión de MEGAN. Los datos de LAI CMAQ los puede levantar de las salidas de MCIP. |
||
| + | |||
| + | 2) Growth form and ecotypes [[https://bai.ess.uci.edu/megan/data-and-code/growth-form-and-ecotypes]]. Existe la base de datos global, pero no está disponible en la página. Están para descargar acá [[https://drive.google.com/file/d/1sCJt66wMd2RY7bSRRDP5DHWL7OB6Va4T/view]] |
||
| + | |||
| + | Y si queremos considerar emisiones de NO del suelo: |
||
| + | |||
| + | 3) Soil NO model inputs [[https://drive.google.com/file/d/1IAnXOpTHKsmXPmAeOiI7o3DyhsKuN7zB/view]]. Se puede descargar directamente de la página. Ojo, acá [[https://forum.cmascenter.org/t/environment-variable-settings-about-biogenic-emissions-in-cmaq/4066]] mencionan que a Abril del 2023 todavía no es recomendable considerar estas emisiones porque no están del todo tuneadas aún. |
||
| + | |||
| + | |||
| + | Lo que se puede descargar, lo vamos a descargar y descomprimir todo en <code> /home/solange.luque/MODELOS/Megan/MEGAN_inputs </code>. |
||
| + | |||
| + | En resumen, lo que necesitamos si o si es los del punto 2: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | |||
| + | cd /home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype |
||
| + | |||
| + | ls |
||
| + | ecotype30s.nc EVT3b.nc GF3aCrop.nc GF3aGrass.nc GF3aShrub.nc GF3aTree.nc NTfrac_reorder_lat.nc tropfrac_reorder_lat.nc |
||
| + | |||
| + | </pre> |
||
| + | |||
| + | ===== Uso ===== |
||
| + | |||
| + | |||
| + | <pre style = "shell"> |
||
| + | /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN32_Prep_Code_Jan_2022 |
||
| + | </pre> |
||
| + | |||
| + | Hay varios programas, pero los que tenemos que correr si o si (el resto son opcionales son): |
||
| + | |||
| + | 1) prepmegan4cmaq_grwform.x: Como input necesita los ficheros GF3 |
||
| + | |||
| + | 2) prepmegan4cmaq_cantype.x: Como input necesita los ficheros GF3, NTfrac y tropfrac |
||
| + | |||
| + | 3) prepmegan4cmaq_ecotype.x: Como input necesita el fichero EVT3 |
||
| + | |||
| + | Cada uno tiene su namelist. Por ejemplo, para mi dominio d01 en primavera, para grwform: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | diff prepmegan4cmaq.growthform.inp prepmegan4cmaq.growthform.inp.old |
||
| + | 3,6c3,6 |
||
| + | < start_lai_mnth = 11, |
||
| + | < end_lai_mnth = 11, |
||
| + | < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', |
||
| + | < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', |
||
| + | --- |
||
| + | > start_lai_mnth = 1, |
||
| + | > end_lai_mnth = 12, |
||
| + | > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', |
||
| + | > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', |
||
| + | 29,32c29,32 |
||
| + | < x0 = 6, |
||
| + | < y0 = 6, |
||
| + | < ncolsin = 77, |
||
| + | < nrowsin = 77, |
||
| + | --- |
||
| + | > x0 = 1, |
||
| + | > y0 = 1, |
||
| + | > ncolsin = 1000, |
||
| + | > nrowsin = 1000, |
||
| + | </pre> |
||
| + | |||
| + | <code>x0</code> y <code>y0</code> son el número de columna y fila respectivamente de la esquina inferior izquierda del dominio de simulación de CMAQ respecto del de WRF. <code>ncolsin</code> y <code>nrowsin</code> son el número de columnas y filas del dominio de CMAQ y se pueden ver fácil de las salidas de MCIP (los ficheros nc y el GRIDDESC). |
||
| + | |||
| + | <code>x0</code> y <code>y0</code> no se ven directo de las salidas, pero sí del log de MCIP. En el log hay que buscar algo de la pinta: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | Output grid resolution: 45.0000000000000 km |
||
| + | Window domain origin on met domain (col,row): 6 , 6 |
||
| + | Window domain far corner on met domain (col,row): 85 , 85 |
||
| + | </pre> |
||
| + | |||
| + | Donde "Window domain origin on met domain (col,row)" es lo que buscamos. Nota respecto de esto: Esto es así porque estamos corriendo CMAQ sacando el borde de WRF. En ese caso, el número de celdas que se sacan son 13 (2*BTRIM+2*NTHIK+1). Si en cambio sacamos manualmente las celdas que queremos, hay que definir un <code>X0</code> e <code>Y0</code> al correr MCIP y esos son los que corresponden a las variables de acá también y se pueden ver en el archivo <code>namelist.mcip</code> (Los valores que están ahí si sacamos automáticamente los valores de los bordes no son los correctos). |
||
| + | |||
| + | Ahora sí, podemos correr. También tenemos un launch (Están todos en <code>/home/solange.luque/estudios/MEGAN_launches</code>). |
||
| + | |||
| + | |||
| + | <pre style = "shell"> |
||
| + | cp /home/solange.luque/estudios/MEGAN_launches/launch_growthform_gnu.pbs . |
||
| + | qsub launch_growthform_gnu.pbs |
||
| + | </pre> |
||
| + | |||
| + | |||
| + | Esto debiera generar el fichero <code>grid_growth_form.csv</code> en la carpeta <code>output</code>. Lo renombramos: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | cd output |
||
| + | mv grid_growth_form.csv grid_growth_form.d01_spring.csv |
||
| + | </pre> |
||
| + | |||
| + | Vamos con cantype: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | diff prepmegan4cmaq.cantype.inp prepmegan4cmaq.cantype.inp.old |
||
| + | 3,6c3,6 |
||
| + | < start_lai_mnth = 11, |
||
| + | < end_lai_mnth = 11, |
||
| + | < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', |
||
| + | < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', |
||
| + | --- |
||
| + | > start_lai_mnth = 1, |
||
| + | > end_lai_mnth = 12, |
||
| + | > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', |
||
| + | > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', |
||
| + | 33,36c33,36 |
||
| + | < x0 = 6, |
||
| + | < y0 = 6, |
||
| + | < ncolsin = 77, |
||
| + | < nrowsin = 77, |
||
| + | --- |
||
| + | > x0 = 1, |
||
| + | > y0 = 1, |
||
| + | > ncolsin = 1000, |
||
| + | > nrowsin = 1000, |
||
| + | |||
| + | </pre> |
||
| + | |||
| + | Corremos: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | qsub launch_cantype_gnu.pbs |
||
| + | </pre> |
||
| + | |||
| + | Esto debería generar el fichero <code>CT3.csv</code> en <code>output</code>. Lo renombramos: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | cd output |
||
| + | mv CT3.csv CT3.d01_spring.csv |
||
| + | </pre> |
||
| + | |||
| + | |||
| + | Finalmente, nos queda por correr el de <code>ecotype</code>. |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | diff prepmegan4cmaq.ecotype.inp prepmegan4cmaq.ecotype.inp.old |
||
| + | 3,6c3,6 |
||
| + | < start_lai_mnth = 11, |
||
| + | < end_lai_mnth = 11, |
||
| + | < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', |
||
| + | < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', |
||
| + | --- |
||
| + | > start_lai_mnth = 1, |
||
| + | > end_lai_mnth = 12, |
||
| + | > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', |
||
| + | > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', |
||
| + | 16,19c16,19 |
||
| + | < x0 = 6, |
||
| + | < y0 = 6, |
||
| + | < ncolsin = 77, |
||
| + | < nrowsin = 77, |
||
| + | --- |
||
| + | > x0 = 1, |
||
| + | > y0 = 1, |
||
| + | > ncolsin = 1000, |
||
| + | > nrowsin = 1000, |
||
| + | </pre> |
||
| + | |||
| + | Y corremos: |
||
| + | |||
| + | |||
| + | <pre style = "shell"> |
||
| + | qsub launch_ecotype_gnu.pbs |
||
| + | </pre> |
||
| + | |||
| + | Esto debería generar el fichero <code>grid_ecotype.csv</code> en <code>output</code>. Lo renombramos: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | cd output |
||
| + | mv grid_ecotype.csv grid_ecotype.d01_spring.csv |
||
</pre> |
</pre> |
||
| Línea 1.292: | Línea 1.496: | ||
Se descarga de acá [[https://drive.google.com/file/d/17K98NWgiZt9J8sl-gF4VEwUXFON-SGZQ/view]]. |
Se descarga de acá [[https://drive.google.com/file/d/17K98NWgiZt9J8sl-gF4VEwUXFON-SGZQ/view]]. |
||
| − | Esto necesita librerías que en el servidor no funcionan bien (como Pandas) por lo que recomendaría correrlo localmente. No solo eso, sino correrlo en un environment. Y no solo eso...sino que está hecho para Python2 así que en principio hay que tener esa versión instalada (No lo probé con python3, quizás con algunas modificaciones funciona). |
+ | Esto necesita librerías que en el servidor no funcionan bien (como Pandas) por lo que recomendaría correrlo localmente. Las librerías que necesita son: |
| + | a)numpy |
||
| + | |||
| + | b)pandas |
||
| + | |||
| + | c)python-dateutil |
||
| + | |||
| + | d)pytz |
||
| + | |||
| + | e)six |
||
| + | |||
| + | |||
| + | Los inputs de este código son las salidas de <code>prep4cmaq_grwform.x</code> y <code>prep4cmaq_ecotype.x</code>. Hay que bajarlas y guardarlas en <code>/home/sol/MEGEFP32/inputs/EFP</code>. |
||
| + | |||
| + | El script a correr es <code>MEGAN_EFP.py</code> en <code>/home/sol/MEGEFP32</code>. En principio, lo único que hay que cambiarle es el nombre del escenario que tiene que coincidir con el sufijo que le pusimos a nuestros inputs. En nuestro caso, <code>d01_spring</code>. |
||
<pre style = "shell"> |
<pre style = "shell"> |
||
| − | virtualenv --python="/usr/bin/python2.7" "Megan_env" |
+ | diff MEGAN_EFP.py MEGAN_EFP.py.old |
| − | source Megan_env/bin/activate |
+ | 39c39 |
| − | cd /home/sol/MEGEFP32 |
+ | < scen_name = "d01_spring" |
| − | pip install numpy |
+ | --- |
| − | pip install pandas |
+ | > scen_name = "tx_12km" |
| − | pip install python-dateutil |
+ | |
| − | pip install pytz |
+ | </pre> |
| − | pip install six |
+ | |
| + | Y corremos: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | python3 MEGAN_EFP.py |
||
</pre> |
</pre> |
||
| − | Las versiones de estas librerías que pide por default en "requirements.txt" no existen más, por lo que puede que haya que modificar algunas cosas (TODAVÍA A CHEQUEAR PORQUE SIN LOS OUTPUTS DEL PRE PROCESADOR DE FORTRAN NO TENGO LOS INPUTS PARA ESTO) |
+ | Si todo sale bien, se genera en <code>/home/sol/MEGEFP32/outputs</code> el fichero <code>OutputGridEF.d01_spring.csv</code> que es el fichero con las emisiones en el dominio. |
| + | |||
| + | |||
| + | Lo subimos al servidor a la carpeta <code>/home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python</code>. |
||
| + | |||
| + | ==== Megan ==== |
||
| − | === Compilación Megan === |
+ | ===== Compilación ===== |
Se descarga de acá [[https://drive.google.com/file/d/1ONtMjwxr9N7bnNDYaaPs1JEt19pvN65P/view]] |
Se descarga de acá [[https://drive.google.com/file/d/1ONtMjwxr9N7bnNDYaaPs1JEt19pvN65P/view]] |
||
| Línea 1.607: | Línea 1.825: | ||
Si no hay ningún error, deberían crearse los ejecutables en cada carpeta de cada programa. |
Si no hay ningún error, deberían crearse los ejecutables en cada carpeta de cada programa. |
||
| + | |||
| + | ===== Uso ===== |
||
| + | |||
| + | Si bien MEGAN tiene varios programas (que son los que compilamos arriba), para usar con CMAQ el único que nos importa es <code>TXT2IOAPI</code> que agarra las salidas de los dos pre procesadores y las convierte de csv a netcdf (CT3.csv de prep4cmaq_cantype y OutputGridEF.csv del pre procesador de Python). |
||
| + | |||
| + | La idea es tener a la salida los siguientes archivo: |
||
| + | |||
| + | 1) CT3.ncf: Este tiene la canopy fraction para seis tipos de vegetación (needleleaf trees, tropical forest trees, temperate broadleaf trees, shrubs, herbaceous plants, y crops) |
||
| + | |||
| + | 2) LDF.ncf: Tiene "light dependence fractions" for 4 of the 19 MEGAN chemical species. |
||
| + | |||
| + | 3) EFMAP.ncf: Factores de emisión para las 19 especies de MEGAN. |
||
| + | |||
| + | |||
| + | Con estos tres archivos, CMAQ ya se encarga de hacer todo. |
||
| + | |||
| + | El script para correr <code>TXT2IOAPI</code> está en: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | cd /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work |
||
| + | </pre> |
||
| + | |||
| + | |||
| + | |||
| + | Lo modificamos: |
||
| + | |||
| + | <pre style = "shell">diff run.txt2ioapi.v32.csh run.txt2ioapi.v32.csh.old |
||
| + | 6c6 |
||
| + | < #foreach dom ( SE_US ) |
||
| + | --- |
||
| + | > foreach dom ( SE_US ) |
||
| + | 10,13c10,12 |
||
| + | < #setenv EXEC $MGNEXE/$PROG |
||
| + | < setenv EXEC /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/src/TXT2IOAPI/txt2ioapi |
||
| + | < setenv GRIDDESC /home/solange.luque/estudios/CMAQ-AMBA/anidados_4_20ppb_20m/d01/spring/DATA/mcip/AMBA_11_d01/GRIDDESC |
||
| + | < setenv GDNAM3D ChemGBsAs_d01_cp |
||
| + | --- |
||
| + | > setenv EXEC $MGNEXE/$PROG |
||
| + | > setenv GRIDDESC $MGNINP/MAP/GRIDDESC |
||
| + | > setenv GDNAM3D $dom |
||
| + | 15c14 |
||
| + | < #set dd = `echo $dom | cut -c 1-2` |
||
| + | --- |
||
| + | > set dd = `echo $dom | cut -c 1-2` |
||
| + | 20c19 |
||
| + | < setenv EFSTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python/OutputGridEF.d01_spring.csv |
||
| + | --- |
||
| + | > setenv EFSTXTF $MGNINP/MAP/OutputGridEF.$dom.csv |
||
| + | 22c21 |
||
| + | < setenv LDFTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python/OutputGridEF.d01_spring.csv |
||
| + | --- |
||
| + | > setenv LDFTXTF $MGNINP/MAP/OutputGridEF.$dom.csv |
||
| + | 24c23 |
||
| + | < setenv CTTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN32_Prep_Code_Jan_2022/output/CT3.d01_spring.csv |
||
| + | --- |
||
| + | > setenv CTTXTF $MGNINP/MAP/CT3.${GDNAM3D}.csv |
||
| + | 28c27 |
||
| + | < setenv LAIv_YN N |
||
| + | --- |
||
| + | > setenv LAIv_YN Y |
||
| + | 44,45c43,44 |
||
| + | < setenv EFMAPS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/EFMAP.d01_spring.ncf |
||
| + | < setenv CANTYPFILE /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/CT3.d01_spring.ncf |
||
| + | --- |
||
| + | > setenv EFMAPS $MGNINP/MAP/EFMAP.2019b.${GDNAM3D}.ncf |
||
| + | > setenv CANTYP $MGNINP/MAP/CT3_${GDNAM3D}.ncf |
||
| + | 48c47 |
||
| + | < setenv LDFILE /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/LDF.d01_spring.ncf |
||
| + | --- |
||
| + | > setenv LDFILE $MGNINP/MAP/LDF_${GDNAM3D}.2019b.ncf |
||
| + | 56c55 |
||
| + | < setenv RUN_EFS T # [T|F] |
||
| + | --- |
||
| + | > setenv RUN_EFS F # [T|F] |
||
| + | 58c57 |
||
| + | < setenv RUN_CANTYP T # [T|F] |
||
| + | --- |
||
| + | > setenv RUN_CANTYP F # [T|F] |
||
| + | 60c59 |
||
| + | < setenv RUN_LDF T # [T|F] |
||
| + | --- |
||
| + | > setenv RUN_LDF F # [T|F] |
||
| + | 63c62 |
||
| + | < setenv RUN_FERT F # [T|F] fertilizer data |
||
| + | --- |
||
| + | > setenv RUN_FERT T # [T|F] fertilizer data |
||
| + | 88c87 |
||
| + | < #end |
||
| + | --- |
||
| + | > end |
||
| + | |||
| + | </pre> |
||
| + | |||
| + | Lo corremos: |
||
| + | |||
| + | <pre style ="shell"> |
||
| + | qsub launch_txt2ioapi_gnu.pbs |
||
| + | </pre> |
||
| + | |||
| + | Si todo sale bien esto debería generar en la carpeta <code>output</code>: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | ls |
||
| + | CT3.d01_spring.ncf EFMAP.d01_spring.ncf LDF.d01_spring.ncf |
||
| + | </pre> |
||
| + | |||
| + | ==== Acoplado con CMAQ ==== |
||
| + | |||
| + | Las salidas de MEGAN para usar con CMAQ (CT3.ncf, EFMAP.ncf y LDF.ncf) son independientes del tiempo. Es decir, no importa para que período se las corra siempre son las mismas. Esto es porque es CMAQ después el encargado de considerar las condiciones meteorológicas y el LAI del período a considerar. Si no se usara CMAQ; sí se necesitarían usar los otros programas de MEGAN que no usamos que se encargarían de hacer esto mismo. |
||
| + | |||
| + | Lo único que hay que hacer parar correr CMAQ con MEGAN es cambiar la siguiente flag en el <code>run_script</code>. |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | setenv CTM_BIOGEMIS_MG Y |
||
| + | </pre> |
||
| + | |||
| + | Y dar los paths de los archivos anteriores en: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | setenv MEGAN_CTS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/CT3.d01_spring.ncf |
||
| + | setenv MEGAN_EFS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/EFMAP.d01_spring.ncf |
||
| + | setenv MEGAN_LDF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/LDF.d01_spring.ncf |
||
| + | </pre> |
||
| + | |||
| + | Si además queremos que CMAQ genere un fichero con las emisiones tenemos que modificar el CMAQ_Control_DESID.nml con lo siguiente: |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel |
||
| + | cp CMAQ_Control_DESID.nml CMAQ_Control_DESID_megan.nml |
||
| + | |||
| + | diff CMAQ_Control_DESID.nml CMAQ_Control_DESID_megan.nml |
||
| + | 203c203 |
||
| + | < Desid_N_Diag_Rules = 0 ! Exact Number of Diagnostic Rules Below |
||
| + | --- |
||
| + | > Desid_N_Diag_Rules = 1 ! Exact Number of Diagnostic Rules Below |
||
| + | 239a240,248 |
||
| + | > |
||
| + | > ! Emisiones MEGAN |
||
| + | > |
||
| + | > Desid_Diag_Streams_Nml(1,:)= 'MIOG' |
||
| + | > Desid_Diag_Fmt_Nml(1) = '3D' ! Options: LAYER1, COLSUM, 3D |
||
| + | > Desid_Diag_Spec_Nml(1,:) = 'ALL' |
||
| + | > |
||
| + | |||
| + | |||
| + | |||
| + | </pre> |
||
| + | |||
| + | |||
| + | Esto va a generar un fichero <code>CCTM_DESID1_MIOG*</code> con las emisiones. |
||
| + | |||
| + | |||
| + | Y ahora en el run script (línea 333): |
||
| + | |||
| + | <pre style = "shell"> |
||
| + | setenv DESID_CTRL_NML ${BLD}/CMAQ_Control_DESID_megan.nml |
||
| + | </pre> |
||
Última revisión de 15:00 19 ene 2024
Inventarios, condiciones de border e iniciales para CMAQ
Contenido |
[editar] Inventarios
[editar] Propios
[editar] Archivos out
Estos archivos están regrillados en formato ASCII y tienen una fila por punto de malla con, los índices i,j y seguido de la serie temporal de datos (ciclo diario) de las emisiones a ese punto de grilla
[j] [i] [emis_t1] ... [emis_tN]
Se tienen las lat,lon de un par de puntos de malla para poder georeferenciar. Se asume que la grilla de datos está en una proyección lat/lon regular (o que la regilla es suficientemente pequeña como para que no se note la curvatura terrestre)
Creamos un python para transformar los ficheros a netCDF
Archivo:out to nc.odt (NOTA: descargar y cambiarle la extensión a .py)
[editar] out_to_nc.py
Sólo requiere las liberías netcdf4-python3. Se usa:
python3 out_to_nc.py -f [archivoout] -v [nombreVariable] -s [nombreStandardVariable]
-l [nombreLargoVariable ('!' para espacios)] -u [unidadesVariable]
-p [iref1],[jref1],[lonref1],[latref2]:[iref2],[jref2],[lonref2],[latref2]
Tenemos dos archivos:
* NOxAMBAtot.out * VOCAMBAtot.out
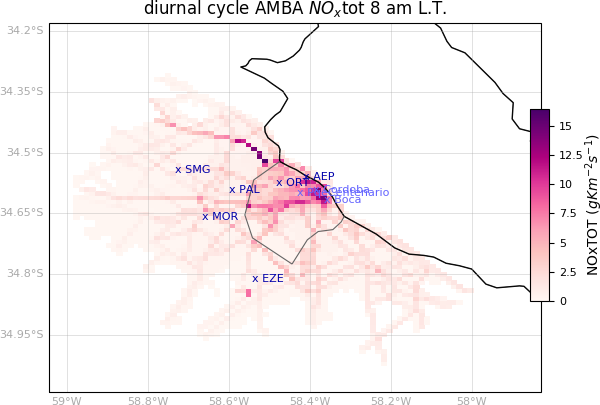 |
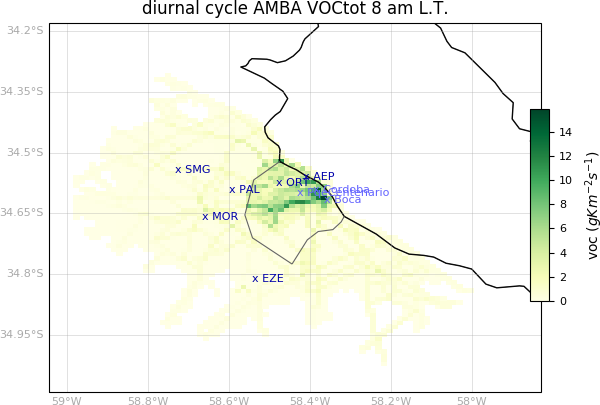
|
Se tienen que preparar como nopoint emisiones del sistema SMOKE. Esto se hace desde el script the python nc_to_nopoint.odt
[editar] nc_to_nopoint.py
Script the python para transformar unos datos en netCDF al formato ASCII de nopoint para SMOKE
[editar] Hermes (Emisiones globales)
Si uno no tiene un inventario para el dominio de interés (Por ejemplo, en caso de querer usar condiciones de borde dinámicas y necesitar correr un dominio más grande), una opción es usar Hermes [[1]]. Todo lo que sigue es para Hermesv3 2.1.3.
[editar] Instalación
[editar] Paquetes de Python
Hermes necesita muchos paquetes de python para correr por lo que conviene instalarlo de forma local en vez de en el servidor. Mas aún, conviene crear un environment para no tener problema con las distintas versiones de las librerías. Una forma práctica de hacerlo es tener un txt con todos los paquetes necesarios así se instalan todos juntos. El txt está en el git [[2]].
virtualenv hermes source hermes/bin/activate pip install -r paquetes.txt
NOTA1: Cuidado con instalar un pandas con versión igual a mayor a 2.0 porque el "append" para un dataframe no se usa más a partir de esa versión y sí se usa en los scripts. Si se instalara una versión superior habría que cambiar los scripts. La recomendación sería instalar una versión inferior y listo.
NOTA2: Si se instala un esmpy posterior a 8.4.0 (Abajo se describe como) hay que renombrar algunas funciones y como se importa el paquete. Abajo se describe. Se puede instalar 7.1.0r que es lo mínimo que pide Hermes y no hay que cambiar nada de los scripts, PERO es una versión vieja que se instala de otra forma y tiene otras complicaciones.
NOTA3: Aparentemente no cualquier versión de netCDF4 funciona bien. En particular la 1.6.4 tira segmentation faults (No al importarlo solo uno a mano, pero sí al funcionar con el resto de las librerías). Netcdf4 1.5.6 con ESMF 8.5.0 funciona bien. Otras combinaciones a chequear.
[editar] Otros programas
Además de los paquetes de python, hay otros programas que son necesarios para la utilización de hermes. Están descriptos acá [[3]]
[editar] ESMF
Este hay que buildearlo. De este programa depende esmpy. Es decir antes de instalar esmpy hay que instalar esto. Es muy importante que se buildee con NETCDF y PIO sino algunas funciones de esmpy que necesitamos no van a funcionar.
Hay que descargarlo de acá [[4]].
Lo vamos a guardar en /home/sol/hermesv3_gr/Libraries/esmf .
cd /home/sol/hermesv3_gr/Libraries/esmf/ cd esmf-8.5.0
Hay que declarar varios paths que están descriptos acá [[5]].
export ESMF_DIR=/home/sol/hermesv3_gr/Libraries/esmf/esmf-8.5.0 export ESMF_NETCDF=nc-config export ESMF_COMM=mpich make all make install make installcheck
NOTA: Si la versión que uno tiene de cmake es inferior a 3.20 hay una función que llama esto que es TestBigEndian.csh. Esta función a mi no me funcionaba y me tiraba error con cualquier compilador (Solo corría con mpiuni que no permite usar PIO). Si eso sucede, no encontré otra solución más que upgradear cmake.
NOTA2: Antes de instalar esmpy no está mal chequear que todo este bien.
make run_examples
y
make run_unit_tests
Si esto está bien, ahora hay que instalar esmpy.
cd src cd addon cd esmpy python3 -m pip install .
Listo.
[editar] Instalación y corrección de errores de Hermes
git clone https://earth.bsc.es/gitlab/es/hermesv3_gr.git cd hermesv3_gr python3 setup.py install
Si alguna dependencia está mal con las librerías anteriores, en este proceso se va a quejar.
A partir de la versión 8.4.0 de esmpy se deja de importar como "import ESMF" y se pasa a importar como "import esmpy". Hay que cambiar esto en los códigos si se usa una versión posterior. Además hay que cambiar el nombre de las funciones.
/home/sol/hermes/lib/python3.8/site-packages/hermesv3_gr-2.1.3-py3.8.egg/hermesv3_gr/modules/regrid/regrid_conservative.py /home/sol/hermes/lib/python3.8/site-packages/hermesv3_gr-2.1.3-py3.8.egg/hermesv3_gr/modules/grids/grid.py
Cada vez que aparezca algo como ESMF.Field o ESMF.Regrid o ESMF.StaggerLoc hay que cambiar el ESMF por esmpy.
También hay que definir bien el path al make file de ESMF. Modificamos el fichero:
/home/sol/hermes/lib/python3.8/site-packages/esmpy/interface/loadESMF.py
Hay que cambiar
esmfmk = os.environ["ESMFMKFILE"]
Por el path del makefile. En mi caso:
esmfmk = "/home/sol/hermesv3_gr/Libraries/esmf/esmf-8.5.0/lib/libO/Linux.gfortran.64.mpich.default/esmf.mk"
También hay que cambiar un par de cosas numpy. A partir de la versión 1.20 no se usa más "np.float" sino directamente "float", salvo que se use np.float32 o np.float64 (idem con int) (Ver la tabla de acá [[6]]). Se podría instalar una versión anterior, pero parece que es vieja y no se puede instalar bien. Lo más directo parece ser cambiar este tipo de cosas en el siguiente script, que es el único con problemas:
/home/sol/hermes/lib/python3.8/site-packages/hermesv3_gr-2.1.3-py3.8.egg/hermesv3_gr/modules/writing/writer_cmaq.py
[editar] Preparación de inventarios
Lo siguiente que hay que hacer es copiar las entradas de muestra:
hermesv3_gr_copy_config_files '/home/sol/HERMES/HERMES_IN'
Acá van a estar todos los ficheros para hacer las especiaciones y para pasar los inventarios anuales a mensuales, diarios y horarios.
Los inventarios (por ejemplo, el de EDGAR) tienen que ser adaptados para funcionar con Hermes. Para eso se necesitan unos scripts que se consiguen con:
hermesv3_gr_copy_preproc_files '/home/sol/HERMES/preproc'
Hay varias opciones de inventarios con sus scripts correspondientes para ser adaptados. Los inventarios disponibles están acá [[7]].
[editar] EDGARv432_AP y EDGARv432_VOC
Estos inventarios los podemos usar y acá abajo se describe como descargarlos y prepararlos. El problema es que en los ficheros de muestra de Hermes no están sus especiaciones con los sectores de estos inventarios. Si uno quisiera, se podría hacer la especiación pero requiere saber como es para cada sector y especie.
[editar] VOCs
Vamos a empezar con el de EDGAR. Lo primero que tenemos que hacer es descargar los inventarios. Esto se hace con el script download_voc_edgar.py que está en GIT [[8]]. Este script guarda todo en el disco externo en una carpeta edgardv432_voc/original_files porque es pesado. El script está hecho para descargar los datos anuales para el 2012, pero se puede cambiar el año.
python3 download_voc_edgar.py
Una vez los inventarios descargados, usamos /home/sol/HERMES/preproc/edgarv432_voc_preproc.py para procesar los inventarios y tengan el formato necesario para Hermes. Tenemos que cambiar los paths:
INPUT_PATH = '/media/sol/External/edgarv432_voc/original_files/'
OUTPUT_PATH = '/media/sol/External/Inventarios_Edgar/edgarv432_voc/'
LIST_POLLUTANTS = ['voc1', 'voc2', 'voc3', 'voc4', 'voc5', 'voc6', 'voc7', 'voc8', 'voc9', 'voc10', 'voc11', 'voc12',
'voc13', 'voc14', 'voc15', 'voc16', 'voc17', 'voc18', 'voc19', 'voc20', 'voc21', 'voc22', 'voc23',
'voc24', 'voc25']
# list_years = [1970, 1971, 1972, 1973, 1974, 1975, 1976, 1977, 1978, 1979, 1980, 1981, 1982, 1983, 1984, 1985, 1986,
# 1987, 1988, 1989, 1990, 1991, 1992, 1993, 1994, 1995, 1996, 1997, 1998, 1999, 2000, 2001, 2002, 2003,
# 2004, 2005, 2006, 2007, 2008, 2009, 2010, 2011, 2012]
LIST_YEARS = [2012]
# To do yearly emissions
PROCESS_YEARLY = True
YEARLY_INPUT_NAME = 'yearly/v432_VOC_spec_<pollutant>_<year>_<ipcc>.0.1x0.1.nc'
# To process monthly emissions, 2010 directly from monthly_input_name and other years calculated using monthly gridded factors derived from the 2010 monthly data
PROCESS_MONTHLY = False
MONTHLY_INPUT_NAME = 'monthly/v432_VOC_spec_<pollutant>_2010_<month>_<ipcc>.0.1x0.1.nc'
MONTHLY_PATTERN_FILE = 'temporal_profiles/v432_FM_<sector>.0.1x0.1.nc'
Además, hay un error de tipeo en la línea 189. Hay que cambiar
nc_var.coordinates = data_atts['coordiantes']
por
nc_var.coordinates = data_atts['coordinates']
Ahora sí. Se corre
python3 edgarv432_voc_preproc.py
Esto tarda en correr, pero genera en el disco externo la carpeta edgarv432_voc/yearly_mean con todos los ficheros en el formato adecuado para Hermes.
[editar] Gases y Aerosoles
Ahora vamos a hacer los mismo, pero para descargar y preparar el inventario de gases y aerosoles que tienen ["BC","CO","NH3","NMVOC","NOx","OC","PM10","PM2.5","SO2"]. Para eso, vamos a usar el script download_ap_edgar.py que está en GIT [[9]]. Nuevamente, esto descarga todo en el disco externo en la carpeta edgardv61_ap/original_files. Está hecho para el año 2012, pero se puede cambiar.
python3 download_ap_edgar.py
Ahora vamos a tener que procesar los inventarios. Como el inventario de Edgar que está ahora en la página es v6.1 en vez de v432 vamos a tener que hacer otro script, en particular porque son otros los sectores que hay en estas versión. Nos copiamos el de la versión anterior y lo modificamos.
cp edgarv432_ap_preproc.py edgarv61_ap_preproc.py
Aparte de cambiarle los paths hay que cambiar algunas cosas más. En la versión 432 de Edgar el nombre de los ficheros tenía el código del sector en vez del nombre como ahora, así que hay que cambiar eso para que los pueda leer bien. Esto funciona ahora para los inventarios anuales (Para 2012 no hay mensuales). Si quisiéramos hacer mensuales, habría que arreglar la función do_monthly_transformation también (La parte donde pone in_pollutant = None si no es alguno de los PM2.5)
diff edgarv61_ap_preproc.py edgarv432_ap_preproc.py
37,39c37
< LIST_POLLUTANTS = ['BC', 'CO', 'NH3', 'NOx', 'OC', 'PM10', 'PM2.5', 'SO2', 'NMVOC']
<
< #LIST_POLLUTANTS = ['PM2.5_bio', 'PM2.5_fossil']
---
> LIST_POLLUTANTS = ['PM2.5_bio', 'PM2.5_fossil']
43c41
< LIST_YEARS = [2012]
---
> LIST_YEARS = [2010]
46,47c44,45
< PROCESS_YEARLY = True
< YEARLY_INPUT_NAME = 'yearly/EDGARv6.1_<pollutant>_<year>_<ipcc>.0.1x0.1.nc'
---
> PROCESS_YEARLY = False
> YEARLY_INPUT_NAME = 'yearly/v432_<pollutant>_<year>_<ipcc>.0.1x0.1.nc'
50c48
< PROCESS_MONTHLY = False
---
> PROCESS_MONTHLY = True
73,75c71
< "IPCC_1A3a_SPS": "TNR_Aviation_SPS",
< "IPCC_1A3b_noRES": "TRO_noRES",
< "IPCC_1A3b_RES": "TRO_RES",
---
> "IPCC_1A3b": "TRO",
83a80
> "IPCC_2D": "FOO_PAP",
86d82
< "IPCC_2D": "FOO_PAP",
87a84
> "IPCC_4C_4D1_4D2_4D4": "AGS",
89d85
< "IPCC_4C_4D": "AGS",
91d86
< "IPCC_6C": "SWD_INC",
92a88
> "IPCC_6C": "SWD_INC",
94d89
<
219c214
< nc_output.setncattr('title', 'EDGARv6.1_AP inventory for the sector {0} and pollutant {1}'.format(
---
> nc_output.setncattr('title', 'EDGARv4.3.2_AP inventory for the sector {0} and pollutant {1}'.format(
223c218
< nc_output.setncattr('source', 'EDGARv6.1_AP', )
---
> nc_output.setncattr('source', 'EDGARv4.3.2_AP', )
241c236
< for ipcc in zip(list(ipcc_to_sector_dict().keys()),list(ipcc_to_sector_dict().values())):
---
> for ipcc in list(ipcc_to_sector_dict().keys()):
245c240
< ipcc[1]))
---
> ipcc))
252,256c247,251
< #if pollutant in ['PM2.5_bio', 'PM2.5_fossil']:
< #in_pollutant = pollutant
< #pollutant = 'PM2.5'
< #else:
< #in_pollutant = None
---
> if pollutant in ['PM2.5_bio', 'PM2.5_fossil']:
> in_pollutant = pollutant
> pollutant = 'PM2.5'
> else:
> in_pollutant = None
267c262
< sector = ipcc_to_sector_dict()[ipcc[0]]
---
> sector = ipcc_to_sector_dict()[ipcc]
269c264
< pollutant = pollutant.replace('.', '')
---
> pollutant = in_pollutant.replace('.', '')
286c281
< pollutant, ipcc[1], file_path))
---
> pollutant, ipcc, file_path))
Esto tarda en correr, pero genera en el disco externo la carpeta edgarv61_ap/yearly_mean con todos los ficheros en el formato adecuado para Hermes.
NOTA: Está para descargar ficheros anuales porque para el 2012 no hay mensuales. Para el 2010 sí hay.
[editar] HTAPv2
La ventaja de este inventario es que su especiación está en el fichero de muestra que viene con Hermes. En la v2 hay datos 2008-2010. En la v3 hay 2000-2018 por lo que quizás convendría usar ese para el 2012, pero el problema es que tiene otros sectores para los cuáles no tenemos la especiación. Conviene seguir con la v2.
La desventaja es que se necesita un fichero con los ratios para especiar los VOCS que estaba en una WIKI que ya no existe más. Se necesitan dos tipos de archivos que amablemente me proporcionaron y están acá [[10]] y acá [[11]].
Además de estos archivos, me compartieron un link con todos los archivos relacionados que estaban en en la WIKI acá [[12]]. Está en el GIT también por las dudas de que el link desaparezca [[13]]
Para descargar el inventario hay un script en git [[14]].
python3 download_HTAP_edgar.py
Esto descarga todo en el disco externo porque es pesado. Está todo en la carpeta Inventarios_Edgar/HTAPv2/original_files.
Una vez descargado, los descomprimo:
cd /media/sol/External/Inventarios_Edgar/HTAPv2/original_files unzip '*.zip'
Ahora vamos a usar el script /home/sol/HERMES/preproc/htapv2_preproc.py para procesarlo.
/home/sol/HERMES/preproc/
Hay que cambiar la parte de los paths.
# ============== CONFIGURATION PARAMETERS ====================== INPUT_PATH = '/media/sol/External/Inventarios_Edgar/HTAPv2/original_files' OUTPUT_PATH = '/media/sol/External/Inventarios_Edgar/HTAPv2/htapv2' INPUT_NAME = 'edgar_HTAP_<pollutant>_emi_<sector>_<year>_<month>.0.1x0.1.nc' INPUT_NAME_AIR = 'edgar_HTAP_emi_<pollutant>_<sector>_<year>.0.1x0.1.nc' INPUT_NAME_SHIPS = 'edgar_HTAP_<pollutant>_emi_SHIPS_<year>.0.1x0.1.nc' # HTAP auxiliary NMVOC emission data for the industry sub-sectors # (http://iek8wikis.iek.fz-juelich.de/HTAPWiki/WP1.1?highlight=%28%28WP1.1%29%29) INPUT_NAME_NMVOC_INDUSTRY = '/media/sol/External/Inventarios_Edgar/HTAPv2/original_files/HTAPv2_NMVOC_INDUSTRY/HTAPv2_NMVOC_<sector>_<year>_<month>.0.1x0.1.nc' # list_years = [2008, 2010] LIST_YEARS = [2010] # RETRO ratios applied to HTAPv2 NMVOC emissions # (http://iek8wikis.iek.fz-juelich.de/HTAPWiki/WP1.1?highlight=%28%28WP1.1%29%29) VOC_RATIO_PATH = '/media/sol/External/Inventarios_Edgar/HTAPv2/original_files/retro_nmvoc_ratio_2000_01x01' VOC_RATIO_NAME = 'retro_nmvoc_ratio_<voc>_2000_0.1deg.nc' VOC_RATIO_AIR_NAME = 'VOC_split_AIR.csv' VOC_RATIO_SHIPS_NAME = 'VOC_split_SHIP.csv'
Notar que VOC_RATIO_PATH y INPUT_NAME_NMVOC_INDUSTRY son los paths de los ficheros que hay que tener aparte. También hay que correr los ficheros "VOC_SPLIT_AIR.csv" y "VOC_SPLIT_SHIP.csv" a la carpeta de los ratios.
Y ahora lo corremos.
NOTA: Si hay poco espacio en el disco, va a haber que liberar algo. Por más que guarde la salida en el disco externo, mientras procesa va ocupando tamaño del disco y si no le alcanza se va a parar. Necesita por lo menos 20 gb.
python3 htapv2_preproc.py
Esto tarda en correr, pero finalmente genera los archivos htapv2/monthly_mean y htapv2/yearly_mean con todos los ficheros procesados.
[editar] Configuración
[editar] Fichero configuración de WRF
Primero tenemos que modificar el fichero con los datos del dominio y la configuración de WRF: HERMES/HERMES_IN/data/global_attributes.csv
cp global_attributes.csv global_attributes.csv.old
Los datos que piden los sacamos de algún fichero de salida de WRF del dominio correspondiente. Por ejemplo, para el dominio intermedio de mi simulación (de 3km) de la configuración p8u1 quedaría:
attribute,value BOTTOM-TOP_GRID_DIMENSION,80 GRIDTYPE,C DIFF_OPT,1 KM_OPT,4 DAMP_OPT,3 DAMPCOEF,0.2 KHDIF,0 KVDIF,0 MP_PHYSICS,8 RA_LW_PHYSICS,4 RA_SW_PHYSICS,4 SF_SFCLAY_PHYSICS,1 SF_SURFACE_PHYSICS,2 BL_PBL_PHYSICS,8 CU_PHYSICS,0 SF_LAKE_PHYSICS,0 SURFACE_INPUT_SOURCE,3 SST_UPDATE,0 GRID_FDDA,0 GFDDA_INTERVAL_M,0 GFDDA_END_H,0 GRID_SFDDA,0 SGFDDA_INTERVAL_M,0 SGFDDA_END_H,0 BOTTOM-TOP_PATCH_START_UNSTAG,1 BOTTOM-TOP_PATCH_END_UNSTAG,79 BOTTOM-TOP_PATCH_START_STAG,1 BOTTOM-TOP_PATCH_END_STAG,80 GRID_ID,2 PARENT_ID,1 I_PARENT_START,43 J_PARENT_START,42 PARENT_GRID_RATIO,5 DT,12 MMINLU,MODIFIED_IGBP_MODIS_NOAH NUM_LAND_CAT,21 ISWATER,17 ISLAKE,21 ISICE,15 ISURBAN,13 ISOILWATER,14 CEN_LAT,-34.46497 CEN_LON,-58.56366
[editar] Fichero de niveles verticales
Tenemos que darle un fichero que diga hasta que altura va cada nivel vertical. Para eso vamos a usar el script vertical_layers_conf.py que está en GIT [[15]]. Para correr este script necesitamos algún fichero de salida de WRF o el output METCRO3D.nc que es salida de MCIP. OJO, NO DAN EXACTAMENTE IGUAL. Con el de CMAQ corre sin quejarse. No probé el de WRF así que recomiendo usar la versión de CMAQ
python3 vertical_layers_conf.py wrf_out_file WRF
o
python3 vertical_layers_conf.py METCROD3D_file CMAQ
Esto va a generar un csv de la pinta CMAQ_*numero de layers*layers_vertical_description.csv
Lo copiamos en la carpeta que corresponde.
cp CMAQ_79layers_vertical_description.csv /home/sol/HERMES/HERMES_IN/data/profiles/vertical/CMAQ_79layers_vertical_description.csv
[editar] Ficheros de perfiles mensuales, semanales, diarios y horarios
Para esto vamos a usar los que ya están por default. Ojo que quizás hay que cambiar el ID de las emisiones.
Están en
/home/sol/HERMES/HERMES_IN/data/profiles/temporal/TemporalProfile_Daily.csv /home/sol/HERMES/HERMES_IN/data/profiles/temporal/TemporalProfile_Hourly.csv /home/sol/HERMES/HERMES_IN/data/profiles/temporal/TemporalProfile_Monthly.csv /home/sol/HERMES/HERMES_IN/data/profiles/temporal/TemporalProfile_Weekly.csv /home/sol/HERMES/HERMES_IN/data/profiles/temporal/tz_world_country_iso3166.csv
El último es un fichero con todos los husos horarios del mundo.
[editar] Fichero de configuración de inventarios
Nos copiamos el original
cd /home/sol/HERMES/HERMES_IN/conf/ cp /home/sol/HERMES/HERMES_IN/conf/EI_configuration.csv EI_configuration.csv.old
La idea de este fichero es decirle que inventarios vamos a usar y para cada especie que perfil de especiación (Para hacer la especiación mensual, la semanal, la diaria, la horaria y la de especies en sí). Cada perfil está asociado a un código que debe coincidir con los códigos de los ficheros de especiaciones. Las columnas están separadas por ";".
Borré los inventarios que no me importan para no meter ruido y me quedé solo con el de HTAPv2.
En la columna "ref_year" va el año del cuál son los inventarios. En la columna "active" va a un 1 si se está usando ese inventario con ese sector y 0 sino. Si la frecuencia es anual, si o si hay que tener perfiles para p_month, p_week y p_hour.
Como el inventario HTAPv2 ya tiene los perfiles asignados no toco nada. Pero si no lo tuviera, hay que elegirlos.
También hay que cambiar los paths donde están los inventarios.
El fichero modificado está en el git [[16]].
[editar] Fichero de especiación
Acá tenemos que crear el fichero de especiación para cb6 aero7 que es el mecanismo que vamos a usar. El fichero que viene por default para Hermes son los de cb5 aero5 y cb5 aero6.
Acá está la tabla con todas las especies [[17]]
Nos copiamos alguno de los que ya están y lo vamos a modificar.
cd /home/sol/HERMES/HERMES_IN/data/profiles/speciation cp Speciation_profile_cb05_aero6_CMAQ.csv Speciation_profile_cb06_aero7_CMAQ.csv
Los códigos de especiación para HAPTV2 son E002-E009. Esa especiación ya está hecha. Solo queda cambiar los nombres de las especies de CMAQ del mecanismo viejo al nuevo.
El fichero modificado está acá [[18]]
[editar] Shapefile con bordes de países
Se necesita descargar de [[19]] el shapefile de la Version 3.6 (Las nuevas no lo tienen en shapefile y no sé si Hermes va a entender otro formato).
cd /home/sol/HERMES/HERMES_IN/data/ mkdir gadm_country_mask cd gadm_country_mask cp /home/sol/Downloads/gadm36_shp/gadm36.shp .
[editar] Fichero pesos moleculares
Usamos el que ya está. Si hay que usar una especie que no esté en la lista hay que agregarla. Está acá:
/home/sol/HERMES/HERMES_IN/data/profiles/speciation/MolecularWeights.csv
[editar] Ejecución
NOTA: Si estamos corriendo un dominio con la misma resolución que un dominio previamente corrido (dx), hay que borrar (o renombrar/correr) las carpetas generadas en:
/home/sol/HERMES/HERMES_IN/data/auxiliar_files
Con el nombre lcc_dx_dx, sino hay errores de compatibilidad.
Para correr Hermes, hay que modificar el fichero de configuración HERMES/HERMES_IN/conf/hermes.conf
cp hermes.conf hermes.conf.old
Modificamos todos los paths.
input_dir = /home/sol/HERMES/HERMES_IN data_path = /media/sol/External/Inventarios_Edgar output_dir = /home/sol/HERMES/HERMES_OUT vertical_description = <input_dir>/data/profiles/vertical/CMAQ_79layers_vertical_description.csv cross_table = <input_dir>/conf/EI_configuration.csv p_vertical = <input_dir>/data/profiles/vertical/Vertical_profile.csv p_month = <input_dir>/data/profiles/temporal/TemporalProfile_Monthly.csv p_week = <input_dir>/data/profiles/temporal/TemporalProfile_Weekly.csv p_day = <input_dir>/data/profiles/temporal/TemporalProfile_Daily.csv p_hour = <input_dir>/data/profiles/temporal/TemporalProfile_Hourly.csv p_speciation = <input_dir>/data/profiles/speciation/Speciation_profile_cb06_aero7_CMAQ.csv molecular_weights = <input_dir>/data/profiles/speciation/MolecularWeights.csv world_info = <input_dir>/data/profiles/temporal/tz_world_country_iso3166.csv countries_shapefile = <input_dir>/data/gadm_country_mask/gadm36.shp
Tener en cuenta que data_path es el path donde están todos los inventarios descargados y preparados. El path por sector se le da recién en el archivo de configuración de emisiones.
Hay que poner datos sobre el dominio que corresponden a los datos del GRIDDESC generado por MCIP. Conviene entonces correr primero MCIP para el dominio de interés y después leer los datos desde ese fichero.
Para una proyección lcc, los datos que piden son estos:
if domain_type == lcc:
lat_1 =
lat_2 =
lon_0 =
lat_0 =
nx =
ny =
inc_x =
inc_y =
x_0 =
y_0 =
La correspondencia con los datos de GRIDDESC es:
if domain_type == lcc:
lat_1 = P_ALP
lat_2 = P_BET
lon_0 = P_GAM
lat_0 = Y_CENT
nx = NCOLS
ny = NROWS
inc_x = XCELL
inc_y = YCELL
x_0 = XORIG
y_0 = YORIG
Que en el griddesc están ordenados así:
GRIDDESC ' ' COORDNAME COORDTYPE P_ALP P_BET P_GAM XCENT YCENT ' ' GRIDNAME COORDNAME XORIG YORIG XCELL YCELL NCOLS NROWS NTHICK ' '
Antes de correr tenemos que modificar unas cositas en el script "/home/sol/hermes/lib/python3.8/site-packages/hermesv3_gr-2.1.3-py3.8.egg/hermesv3_gr/modules/emision_inventories/emission_inventory.py". En la línea 92 tenemos que poner que reference_year es un int.
self.reference_year = int(reference_year)
Y ahora sí. Finalmente ejecutamos.
mpirun -np 8 hermesv3_gr --my-config '/home/sol/HERMES/HERMES_IN/conf/hermes.conf'
Esto tarda en correr, pero debiera ir creando los ficheros de las emisiones en el path de salida que elegimos. No hay que hacerles nada, funcionan así directamente con CMAQ.
[editar] Megan (Emisiones biogénicas)
Megan [[20]] es un modelo que genera ficheros de emisiones biogénicas para los dominios de interés. En principio todos los outputs necesarios para el modelo están en bases de datos públicas y no se necesita nada extra. Hay dos versiones disponibles: 2.1 [[21]]y 3.2[[22]]. En principio 3.2 es mejor para zonas urbanas, pero aparentemente necesita más cosas para funcionar y aún no está del todo cerrado el modelo. Además, requiere muchos inputs de datos que en teoría debieran estar disponibles para descargar, pero no están en la página.
[editar] Megan 2.1
Los códigos están acá [[23]]
[editar] Instalación
Antes de instalar Megan hay que instalar netcdf 3.6.0 [[24]] (o mayor) y ioapi 3.1 [[25]].
Acá hay un detalle: Megan viene por default para ser compilado con pgf90 y es necesario que todas las librerías estén compiladas con el mismo compilador. En mi caso, netcdf y ioapi están compiladas con gfortran por lo que toca cambiar las flags de compilación de Megan. En caso de querer re-compilar las librerías con pgf90 (aunque no lo recomiendo porque a mi no me funcionó) debajo hay instrucciones de como (hasta donde llegué al menos).
Descargamos entonces el código de Megan y lo guardamos en el home. Extraemos y:
cd MEGANv2.10_beta mkdir Libraries mkdir Downloads cd Downloads
[editar] Instalación netcdf (en caso de querer re compilar con pgf90)
Megan 2.1 necesita netcdf 3.6.0 o mayor. Al momento de escribir esta WIKI la versión que está para descargar de netcdf-c es 4.9.2 y de netcdf-fortran es 4.6.1. Descargamos ambas. En la carpeta "Downloads"
mkdir netcdf-c_4_9_2 cd netcdf-c_4_9_2
Ahí extraemos el fichero.
cd netcdf-c-4.9.2 export FC=pgf90 export CPPFLAGS="-fPIC -I/home/sol/Build_CMAQ/LIBRARIES/curl/include" export LDFLAGS="-L/home/ubuntu/Build_CMAQ/LIBRARIES/curl/lib" ./configure --prefix=/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2 --disable-libxml2 --disable-netcdf-4 make -j4 make install
Una vez terminado, para que nos tome esta versión de netcdf y no la que ya teníamos instalada,
export PATH=/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2/bin:$PATH
Podemos chequear que sea la versión correcta
nc-config --version
En este caso netCDF 4.9.2.
NOTA: Si salimos de esta terminal, vamos a tener la versión de netcdf anterior. Siempre que la querramos usar que volver a exportar el path.
Ahora vamos con netcdf-fortran.
cd ../.. mkdir netcdf-fortran_4_6_1 cd netcdf-fortran_4_6_1
Acá extraemos el fichero.
cd netcdf-fortran-4.6.1 export CPPFLAGS="-fPIC -I/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2/include" export LDFLAGS="-L/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2/lib" export LD_LIBRARY_PATH="/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2/lib:$LD_LIBRARY_PATH" ./configure --prefix=/home/sol/MEGANv2.10_beta/Libraries/netcdf-4.9.2 make -j4 make install
En caso de que en algún momento haya que volver a correr el configure o el make, conviene antes hacer un make distclean. A mi esto me tira error y no lo puedo terminar de instalar.
[editar] Cambio de flags para compilar Megan con gfortran
En el caso de no querer re instalar y compilar las librerías, tenemos que adecuar los makefiles de Megan al compilador de interés (en mi caso gfortran). Esto está hecho en el servidor.
cd /home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu/src
Acá hay 5 carpetas
EMPROC IOAPI2UAM MET2MGN MGN2MECH TXT2IOAPI
En todas está el makefile correspondiente. Hay que cambiar el compilador, las flags y poner bien los paths a las librerías.
diff Makefile.emproc.64bit Makefile.emproc.64bit.old 2,11c2,3 < < FC = gfortran < #FFLAGS = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp -DF90=1 < #FFLAGS = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 < FFLAGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < < #FC = ifort < #FFLAGS = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ < # -convert big_endian -assume byterecl -traceback -xHost-fopenmp < --- > FC= pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Mlfs -Bstatic_pgi 14,22c6,9 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ --- > LIBS = -L/data3/home/xjiang/bin/ioapi_3.1/Linux2_x86pg_pgcc_nomp -lioapi \ > -L/usr/local/netcdf-4.1.1/lib -lnetcdf > INCLUDE = -I/data3/home/xjiang/bin/ioapi_3.1/ioapi/fixed_src \ > -I/usr/local/netcdf-4.1.1/include \
diff Makefile.ioapi2uam.64bit.old Makefile.ioapi2uam.64bit 2,3c2,14 < FC = pgf90 < FFLAGS = -O2 -Mdalign -Mnoframe -byteswapio -Mlfs -Bstatic_pgi --- > > FC = gfortran > #FFLAGS = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp -DF90=1 > #FFLAGS = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 > FFLAGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp > > #FC = ifort > #FFLAGS = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ > # -convert big_endian -assume byterecl -traceback -xHost-fopenmp > > #FC = ifort > #FFLAGS = -O3 -fixed -132 -traceback -qopenmp -xHost -Bstatic > 5,7c16,22 < LIBS = -L/data3/home/xjiang/bin/ioapi_3.1/Linux2_x86pg_pgcc_nomp -lioapi \ < -L/usr/local/netcdf-4.1.1/lib -lnetcdf < INCLUDE = -I/data3/home/xjiang/bin/ioapi_3.1/ioapi/fixed_src --- > > LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ > -L//opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ > -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp > > INCLUDE = -I//home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ > -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi
diff Makefile.met2mgn.64bit.old Makefile.met2mgn.64bit 1,4c1,28 < PGF90 = pgf90 < FCFLGS = -O -Mextend -Mlfs -Msecond_underscore < FCFLGS2 = -O -Mbyteswapio -Mextend -Mlfs -Msecond_underscore < LNKFLGS = -Bstatic_pgi --- > > PGF90 = gfortran > > > #FCFLGS = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp FFLAGS -DF90=1 > > #FCFLGS2 = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp FFLAGS -DF90=1 > > #FCFLGS = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 > #FCFLGS2 = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 > > FCFLGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp > FCFLGS2 = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp > > > LNKFLGS = -fopenmp > > > #FC = ifort > #FCFLGS = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ > # -convert big_endian -assume byterecl -traceback -xHost-fopenmp > > #FCLGS2 = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ > # -convert big_endian -assume byterecl -traceback -xHost-fopenmp > > > #LNKFLGS = -fopenmp > 22,24c46,52 < LIBS = -L/data3/home/xjiang/bin/ioapi_3.1/Linux2_x86pg_pgcc_nomp -lioapi \ < -L/usr/local/netcdf-4.1.1/lib -lnetcdf < INC = -I/data3/home/xjiang/bin/ioapi_3.1/ioapi/fixed_src --- > > LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ > -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ > -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp > > INC = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ > -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi
diff Makefile.mgn2mech.64bit.old Makefile.mgn2mech.64bit 1,3c1,14 < SHELL=/bin/sh < FC= pgf90 < FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Mlfs -Bstatic_pgi --- > SHELL=/bin/sh > > FC = gfortran > #FFLAGS = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 > #FFLAGS = -dynamic -L/usr/lib64 -lm -lpthread -lc > #FFLAGS = -ffree-line-length-none > #FC = ifort > #FFLGS = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ > # -convert big_endian -assume byterecl -traceback -xHost-fopenmp > > FFLAGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp > > #FC = ifort > #FFLAGS = -O3 -fixed -132 -traceback -qopenmp -xHost -Bstatic 6,9c17,23 < LIBS = -L/data3/home/xjiang/bin/ioapi_3.1/Linux2_x86pg_pgcc_nomp -lioapi \ < -L/usr/local/netcdf-4.1.1/lib -lnetcdf < INCLUDE = -I/data3/home/xjiang/bin/ioapi_3.1/ioapi/fixed_src \ < -I/usr/local/netcdf-4.1.1/include \ --- > LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ > -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ > -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp > > INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ > -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ > -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \
diff Makefile.txt2ioapi.64bit.old Makefile.txt2ioapi.64bit 2,3c2,14 < FC = pgf90 < FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs --- > > FC = gfortran > #FFLAGS = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp -DF90=1 > #FFLAGS = -O3 -ffixed-line-length-132 -fopenmp -ffast-math -funroll-loops -m64 > FFLAGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp > #FC = ifort > #FFLGS = -O3 -extend-source 132 -unroll -stack_temps -safe_cray_ptr \ > # -convert big_endian -assume byterecl -traceback -xHost-fopenmp > > > #FC = ifort > #FFLAGS = -O3 -fixed -132 -traceback -qopenmp -xHost -Bstatic > 5,8c16,22 < LIBS = -L/data3/home/xjiang/bin/ioapi_3.1/Linux2_x86pg_pgcc_nomp -lioapi \ < -L/usr/local/netcdf-4.1.1/lib -lnetcdf < INCLUDE = -I/data3/home/xjiang/bin/ioapi_3.1/ioapi/fixed_src \ < -I/usr/local/netcdf-4.1.1/include \ --- > LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ > -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ > -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp > > INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ > -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ > -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \
Ahora necesitamos cambiar unas cositas de formato en algunos scripts.
cd IOAPI2UAM diff ioapi2uam.f ioapi2uam.f.old 165c165 < write(*,'(2(i9,f10.2))') ibdate,btime,iedate,etime --- > write(*,'(2(i,f))') ibdate,btime,iedate,etime 299c299 < write(*,'(a,2(i9,f10.2))')'Writing ',ibdate,btime,iedate,etime --- > write(*,'(a,2(i,f))')'Writing ',ibdate,btime,iedate,etime
cd ../MET2MGN diff readmcip.f readmcip.f.old 34c34 < logical MCIP_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil,lfirst --- > logical MCIP_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil 383c383 < if(ifsoil) then --- > if(isoil) then diff readpar.f readpar.f.old 15,20c15,16 < INTEGER, parameter :: mx=350 < INTEGER, parameter :: my=410 < INTEGER, parameter :: mz=30 < INTEGER, parameter :: nlat=61 < INTEGER, parameter :: nlon=121 < --- > parameter mx=350,my=410,mz=30 > parameter nlat=61,nlon=121
cd ../TXT2IOAPI diff cnvt_efs.F cnvt_efs.F.old 51c51 < INTEGER :: XREF(MXTCOL) --- > INTEGER, XREF(MXTCOL) diff cnvt_pft.F cnvt_pft.F.old 55c55 < INTEGER :: XREF(NFLD) --- > INTEGER, XREF(NFLD) diff cnvt_lai.F cnvt_lai.F.old 55c55 < INTEGER :: XREF(NFLD) --- > INTEGER, XREF(NFLD)
cd ../EMPROC/INCLDIR diff CONST_MEGAN.EXT CONST_MEGAN.EXT.old 28c28 < & 0.756, 0.756 , 1. , 1. , --- > & 0.756, 0.756 , 1. , 1 , 37,40c37,40 < & 24. , 24. , 24. , 24. , < & 24. , 24. , 24. , 24. , < & 2. , 2. , 2. , 0.75 , < & 0.75 , 0.75 , 1. , 1. , --- > & 24. , 24. , 24 , 24 , > & 24. , 24. , 24 , 24 , > & 2. , 2 , 2 , 0.75 , > & 0.75 , 0.75 , 1. , 1 ,
En ../MGN2MECH/INCLDIR/EFS_PFT.EXT hay que hacer lo mismo: Escribir como floats los integers (Por ejemplo: Si dice 200 hay que poner 200.)
[editar] Cambios configuración y compilación
Hay que cambiar unos paths en los siguientes ficheros:
/home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu/setcase.csh
Ahí hay que cambiar el MGNHOME por /home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu.
En:
/home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu/src/make_all_programs.scr
Hay que cambiar path que sourcea al fichero anterior de setcase.csh. Es decir:
source /home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu/setcase.csh
Y ahora se corre:
cd /home/solange.luque/MODELOS/Megan/MEGANv2.10_gnu/src source /opt/load-libs.sh 4 ./make_all_programs.scr 64bit &> make_gnu.log
Hay que chequear el log por errores, pero con los cambios anteriores no debiera haber problema (Sí hay algunos warnings, pero no importa). Tendrían que aparecen los ejecutables txt2ioapi,
met2mgn, emproc, mgn2mech, y ioapi2uam en cada una de las carpetas.
[editar] Preprocessor tool
Es necesario mapear la data a los dominios de simulación. Esto no lo hace Megan, sino que lo hace un preprocesador que se descarga del punto 3 de esta lista [[26]]. Lo descargamos en:
cd /home/solange.luque/MODELOS/Megan tar -xvzf prepmegan4cmaq_2014-06-02.tar.gz
En principio los programas del preprocesador ya vienen compilados para Linux:
prepmegan4cmaq_ef.x: Necesita datos de global emissions factors (Punto 6 de la lista) prepmegan4cmaq_lai.x: Necesita los datos de LAI (Leaf Area index) (Punto 4 de la lista) prepmegan4cmaq_pft.x: Necesita datos de PFT (Plant Functional Type, aunque esto creo solo está para EEUU, chequear) (Punto 5 de la lista)
[editar] Datasets
Acá hay un tema. Estos son los datasets de la página:
LAI [[27]] PFT [[28]] EF [[29]]
Pero, LAI y PFT solo están para Norte America. Sí podemos descargar y usar los de EF:
Los vamos a guardar en /home/solange.luque/MODELOS/Megan/MEGAN_inputs.
cd /home/solange.luque/MODELOS/Megan/MEGAN_v2.10_inputs unzip GlobalEmissionFactor-20231222T163258Z-001.zip cd GlobalEmissionFactor/ gzip -d *.nc.gz
Descomprimir los archivos dentro de las carpetas puede llevar tiempo porque algunos son globales y hay muchas lat/lon.
PARA LAI y PFT toca descargar las imágenes satelitales de MODIS y hacer uno los ficheros.
[editar] Megan 3.2
Megan 3.2 [[30]] tiene dos pre procesadores. En total son 3 códigos a bajar: Un pre procesador de Fortran (Que agarra los datasets y los mapea al dominio de simulación), el pre procesador de Python (Que Calcula los factores de emisión) y Megan propiamente dicho.
[editar] Pre procesador de Fortran
[editar] Compilación
Se descarga de acá [[31]] y acá [[32]]
Descargamos acá:
/home/solange.luque/MODELOS/Megan mkdir MEGAN3.2_gnu cd MEGAN3.2_gnu tar -xvz MEGAN32_Prep_Code_Jan_2022.tar.gz tar -xvf MEGAN32_Prep_Code_Jan_2022.tar.gz MEGAN32_Prep_Code_Jan_2022 cd MEGAN32_Prep_Code_Jan_2022
Acá tenemos que editar los ficheros make_util y Makefile.
Como mis librerías están compiladas con gfortran, en make_util:
diff make_util make_util.old 4c4 < setenv FC gfortran --- > setenv FC ifort 11c11 < setenv NETCDF_DIR /opt/netcdf/netcdf-4/gnu/10.2.1/ --- > setenv NETCDF_DIR $NETCDF
y en Makefile
diff Makefile Makefile.old 3,4c3,4 < F90 = gfortran #$(FC) < #LIBS = -L$(NETCDF_DIR)/lib -lnetcdf -lnetcdff --- > F90 = ifort #$(FC) > #LIBS = -L$(NETCDF_DIR)/lib -lnetcdf 7c7 < FFLAGS = -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FFLAGS = -O3 -Bstatic
Hay que hacer una modificación en el fichero prepmegan4cmaq_lai.f90.
diff prepmegan4cmaq_lai.f90.old prepmegan4cmaq_lai.f90 831c831 < write (99, '(3(I,","), 48(F10.4,","))') & --- > write (99, '(3(I9,","), 48(F10.4,","))') &
Y en prepmegan4cmaq_cantype.f90.
diff prepmegan4cmaq_cantype.f90 prepmegan4cmaq_cantype.f90.old 190,191c190,191 < missing_value = -128 < !read(treefillvalue,"(f7.0)") missing_value --- > !missing_value = -1. > read(treefillvalue,"(f7.0)") missing_value 195,196c195 < missing_value = 0. < !read(NTfillvalue,"(f7.0)") missing_value --- > read(NTfillvalue,"(f7.0)") missing_value 202,203c201 < missing_value = -1 < !read(TRTfillvalue,"(f7.0)") missing_value --- > read(TRTfillvalue,"(f7.0)") missing_value 206d203 < missing_value = -128 209c206 < !read(shrubfillvalue,"(f7.0)") missing_value --- > read(shrubfillvalue,"(f7.0)") missing_value 213c210 < !read(cropfillvalue,"(f7.0)") missing_value --- > read(cropfillvalue,"(f7.0)") missing_value 217c214 < !read(grassfillvalue,"(f7.0)") missing_value --- > read(grassfillvalue,"(f7.0)") missing_value 982c979 < write (99, '(3(I0,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, & --- > write (99, '(3(I,","), 6(F10.4,","))')cell_id,ilon_mcip,ilat_mcip, &
Ojo, acá también le estoy harcodeando los missing_values para cada archivo. En caso de cambiar, hay que cambiarlos.
Y en prepmegan4cmaq_w126.f90.
diff prepmegan4cmaq_w126.f90 prepmegan4cmaq_w126.f90.old 781c781 < write (99, '(3(I9,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, & --- > write (99, '(3(I,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, &
Y en prepmegan4cmaq_grwform.f90
diff prepmegan4cmaq_grwform.f90 prepmegan4cmaq_grwform.f90.old 210,211c210,211 < missing_value = -128 < !read(treefillvalue,"(f7.0)") missing_value --- > !missing_value = -1. > read(treefillvalue,"(f7.0)") missing_value 215c215 < !read(shrubfillvalue,"(f7.0)") missing_value --- > read(shrubfillvalue,"(f7.0)") missing_value 219c219 < !read(cropfillvalue,"(f7.0)") missing_value --- > read(cropfillvalue,"(f7.0)") missing_value 223c223 < !read(grassfillvalue,"(f7.0)") missing_value --- > read(grassfillvalue,"(f7.0)") missing_value
Ojo con este: Le setee -128 como missing_value porque es el que tienen los ficheros que voy a usar (todos el mismo). Si no fuera el caso, habría que leerlo de los ficheros como estaba antes, pero por algún motivo no funcionaba.
Y ahora corremos:
./make_util prepmegan4cmaq_lai.x ./make_util prepmegan4cmaq_grwform.x ./make_util prepmegan4cmaq_ecotype.x ./make_util prepmegan4cmaq_cantype.x ./make_util prepmegan4cmaq_w126.x
Si además queremos considerar emisiones de NO de suelo, tenemos que compilar más códigos. Hay que modificarlos también:
diff prepmegan4cmaq_arid.f90 prepmegan4cmaq_arid.f90.old 787c787 < write (99, '(3(I9,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, & --- > write (99, '(3(I,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, &
diff prepmegan4cmaq_non_arid.f90 prepmegan4cmaq_non_arid.f90.old 787c787 < write (99, '(3(I9,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, & --- > write (99, '(3(I,","), F10.4,",")')cell_id,ilon_mcip,ilat_mcip, &
diff prepmegan4cmaq_fert.f90 prepmegan4cmaq_fert.f90.old 804c804 < write (99, '(3(I9,","), 2(F10.4,","), 366(E10.4,","))') & --- > write (99, '(3(I,","), 2(F10.4,","), 366(E10.4,","))') &
diff prepmegan4cmaq_landtype.f90 prepmegan4cmaq_landtype.f90.old 799c799 < write (99, '(3(I9,","), 2(F10.4,","),1(I9,","))') & --- > write (99, '(3(I,","), 2(F10.4,","),1(I,","))') &
diff prepmegan4cmaq_nitrogen.f90 prepmegan4cmaq_nitrogen.f90.old 770,771c770,771 < write(99,'(a)') "CELL_ID,X,Y,LAT,LONG,NITROGEN01,NITROGEN02,NITROGEN03,NITROGEN04,NITROGEN05,NITROGEN06, & < NITROGEN07,NITROGEGEN08,NITROGEN09,NITROGEN10,NITROGEN11,NITROGEN12" --- > write(99,'(a)')"CELL_ID,X,Y,LAT,LONG,NITROGEN01,NITROGEN02,NITROGEN03,NITROGEN04,NITROGEN05,NITROGEN06,NITROGEN07,NITROGEN08,NITROGEN09,NITROGEN10, & > NITROGEN11,NITROGEN12" 795c795 < write (99, '(3(I9,","), 2(F10.4,","),12(E14.6,","))') & --- > write (99, '(3(I,","), 2(F10.4,","),12(E14.6,","))') &
Y ahora sí buildeamos:
./make_util prepmegan4cmaq_arid.x ./make_util prepmegan4cmaq_non_arid.x ./make_util prepmegan4cmaq_fert.x ./make_util prepmegan4cmaq_landtype.x ./make_util prepmegan4cmaq_nitrogen.x
[editar] Inputs
El pre procesador de Fortran necesita varios inputs:
1) Leaf Index Area [[33]]. Hay una versión global, pero solo sirve para Megan2. Tiene otro formato (no está en netcdf). En cambio, lo que se puede hacer es generarse los netcdfs necesarios con datos de MODIS. De todas formas, no son obligatorios para usar esta versión de MEGAN. Los datos de LAI CMAQ los puede levantar de las salidas de MCIP.
2) Growth form and ecotypes [[34]]. Existe la base de datos global, pero no está disponible en la página. Están para descargar acá [[35]]
Y si queremos considerar emisiones de NO del suelo:
3) Soil NO model inputs [[36]]. Se puede descargar directamente de la página. Ojo, acá [[37]] mencionan que a Abril del 2023 todavía no es recomendable considerar estas emisiones porque no están del todo tuneadas aún.
Lo que se puede descargar, lo vamos a descargar y descomprimir todo en /home/solange.luque/MODELOS/Megan/MEGAN_inputs .
En resumen, lo que necesitamos si o si es los del punto 2:
cd /home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype ls ecotype30s.nc EVT3b.nc GF3aCrop.nc GF3aGrass.nc GF3aShrub.nc GF3aTree.nc NTfrac_reorder_lat.nc tropfrac_reorder_lat.nc
[editar] Uso
/home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN32_Prep_Code_Jan_2022
Hay varios programas, pero los que tenemos que correr si o si (el resto son opcionales son):
1) prepmegan4cmaq_grwform.x: Como input necesita los ficheros GF3
2) prepmegan4cmaq_cantype.x: Como input necesita los ficheros GF3, NTfrac y tropfrac
3) prepmegan4cmaq_ecotype.x: Como input necesita el fichero EVT3
Cada uno tiene su namelist. Por ejemplo, para mi dominio d01 en primavera, para grwform:
diff prepmegan4cmaq.growthform.inp prepmegan4cmaq.growthform.inp.old 3,6c3,6 < start_lai_mnth = 11, < end_lai_mnth = 11, < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', --- > start_lai_mnth = 1, > end_lai_mnth = 12, > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', 29,32c29,32 < x0 = 6, < y0 = 6, < ncolsin = 77, < nrowsin = 77, --- > x0 = 1, > y0 = 1, > ncolsin = 1000, > nrowsin = 1000,
x0 y y0 son el número de columna y fila respectivamente de la esquina inferior izquierda del dominio de simulación de CMAQ respecto del de WRF. ncolsin y nrowsin son el número de columnas y filas del dominio de CMAQ y se pueden ver fácil de las salidas de MCIP (los ficheros nc y el GRIDDESC).
x0 y y0 no se ven directo de las salidas, pero sí del log de MCIP. En el log hay que buscar algo de la pinta:
Output grid resolution: 45.0000000000000 km Window domain origin on met domain (col,row): 6 , 6 Window domain far corner on met domain (col,row): 85 , 85
Donde "Window domain origin on met domain (col,row)" es lo que buscamos. Nota respecto de esto: Esto es así porque estamos corriendo CMAQ sacando el borde de WRF. En ese caso, el número de celdas que se sacan son 13 (2*BTRIM+2*NTHIK+1). Si en cambio sacamos manualmente las celdas que queremos, hay que definir un X0 e Y0 al correr MCIP y esos son los que corresponden a las variables de acá también y se pueden ver en el archivo namelist.mcip (Los valores que están ahí si sacamos automáticamente los valores de los bordes no son los correctos).
Ahora sí, podemos correr. También tenemos un launch (Están todos en /home/solange.luque/estudios/MEGAN_launches).
cp /home/solange.luque/estudios/MEGAN_launches/launch_growthform_gnu.pbs . qsub launch_growthform_gnu.pbs
Esto debiera generar el fichero grid_growth_form.csv en la carpeta output. Lo renombramos:
cd output mv grid_growth_form.csv grid_growth_form.d01_spring.csv
Vamos con cantype:
diff prepmegan4cmaq.cantype.inp prepmegan4cmaq.cantype.inp.old 3,6c3,6 < start_lai_mnth = 11, < end_lai_mnth = 11, < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', --- > start_lai_mnth = 1, > end_lai_mnth = 12, > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', 33,36c33,36 < x0 = 6, < y0 = 6, < ncolsin = 77, < nrowsin = 77, --- > x0 = 1, > y0 = 1, > ncolsin = 1000, > nrowsin = 1000,
Corremos:
qsub launch_cantype_gnu.pbs
Esto debería generar el fichero CT3.csv en output. Lo renombramos:
cd output mv CT3.csv CT3.d01_spring.csv
Finalmente, nos queda por correr el de ecotype.
diff prepmegan4cmaq.ecotype.inp prepmegan4cmaq.ecotype.inp.old 3,6c3,6 < start_lai_mnth = 11, < end_lai_mnth = 11, < wrffile = '/home/solange.luque/estudios/WRF_new_sims_4_2_1/4_domains_20m/spring/real/outwrf/wrfout_d01_20m_total.nc', < megan_dir = '/home/solange.luque/MODELOS/Megan/MEGAN_inputs/MEGANv3.2/Growth_form_ecotype', --- > start_lai_mnth = 1, > end_lai_mnth = 12, > wrffile = './input/wrfout_d02_2018-05-01_00:00:00', > megan_dir = '/glade/u/home/huiw16/work/MEGAN/MEGAN31_Prep_test_July', 16,19c16,19 < x0 = 6, < y0 = 6, < ncolsin = 77, < nrowsin = 77, --- > x0 = 1, > y0 = 1, > ncolsin = 1000, > nrowsin = 1000,
Y corremos:
qsub launch_ecotype_gnu.pbs
Esto debería generar el fichero grid_ecotype.csv en output. Lo renombramos:
cd output mv grid_ecotype.csv grid_ecotype.d01_spring.csv
[editar] Pre procesador de Python
Se descarga de acá [[38]].
Esto necesita librerías que en el servidor no funcionan bien (como Pandas) por lo que recomendaría correrlo localmente. Las librerías que necesita son:
a)numpy
b)pandas
c)python-dateutil
d)pytz
e)six
Los inputs de este código son las salidas de prep4cmaq_grwform.x y prep4cmaq_ecotype.x. Hay que bajarlas y guardarlas en /home/sol/MEGEFP32/inputs/EFP.
El script a correr es MEGAN_EFP.py en /home/sol/MEGEFP32. En principio, lo único que hay que cambiarle es el nombre del escenario que tiene que coincidir con el sufijo que le pusimos a nuestros inputs. En nuestro caso, d01_spring.
diff MEGAN_EFP.py MEGAN_EFP.py.old 39c39 < scen_name = "d01_spring" --- > scen_name = "tx_12km"
Y corremos:
python3 MEGAN_EFP.py
Si todo sale bien, se genera en /home/sol/MEGEFP32/outputs el fichero OutputGridEF.d01_spring.csv que es el fichero con las emisiones en el dominio.
Lo subimos al servidor a la carpeta /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python.
[editar] Megan
[editar] Compilación
Se descarga de acá [[39]]
Esto sí lo volvemos a correr en el servidor. Lo descargamos acá:
cd /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu unzip MEGANv3.2_Aug_2022.zip cd MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/
Acá cambiamos el path del megan home en setcase.csh.
diff setcase.csh setcase.csh.old 2c2 < setenv MGNHOME "/home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021" --- > setenv MGNHOME "/glade/u/home/huiw16/work/MEGAN/MEGAN32_1227_2021/MEGANv3.2_Dec_2021"
Ahora tenemos que cambiar los makefiles. En el servidor las librerías están compiladas con gfortran así que usamos ese compilador para Megan.
cd src/DAYMET diff makefile makefile.pgf90 2,4c2,3 < FC= gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < --- > FC= pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 6,15c5,8 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ < -I$(CURDIR)/INCLDIR < --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I//usr/local/netcdf-4.7.0/include
cd ../IOAPI2UAM diff makefile makefile.pgf90 2,3c2,3 < FC = gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FC = pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 7,15c7,10 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ < -I$(CURDIR)/INCLDIR --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I/usr/local/netcdf-4.7.0/include
cd ../MEGCAN diff makefile makefile.pgf90 2,3c2,3 < FC = gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FC = pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 7,16c7,10 < < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ < -I$(CURDIR)/INCLDIR --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I/usr/local/netcdf-4.7.0/include
cd ../MEGSEA diff makefile makefile.pgf90 2,3c2,3 < FC = gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FC = pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 7,14c7,10 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I/usr/local/netcdf-4.7.0/include \ 15a12 > 15a12 >
cd ../MEGVEA diff makefile makefile.pgf90 2,3c2,3 < FC = gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FC = pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 7,15c7,10 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ < -I$(CURDIR)/INCLDIR --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I/usr/local/netcdf-4.7.0/include
cd ../MET2MGN diff makefile makefile.pgf90 4,8c4,8 < PGF90 = gfortran < FCFLGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < FCFLGS2 = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < FCFLGS3 = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < LNKFLGS = -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > PGF90 = pgf90 > FCFLGS = -O -Mextend -Mlfs -Msecond_underscore > FCFLGS2 = -O -Mbyteswapio -Mextend -Mlfs -Msecond_underscore > FCFLGS3 = -O2 -Mnoframe -Mextend -Bstatic_pgi > LNKFLGS = -Bstatic_pgi 10,17c10,13 < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INC = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi < -I$(CURDIR)/INCLDIR --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INC = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I/usr/local/netcdf-4.7.0/include
cd ../MGN2MECH diff makefile makefile.pgf90 2,3c2,3 < FC= gfortran < FFLAGS= -O3 -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp --- > FC= pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 5,12c5,8 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I//usr/local/netcdf-4.7.0/include \
cd ../TXT2IOAPI diff makefile makefile.pgf90 4,6c4,5 < FC = gfortran < FFLAGS= -O3 -fallow-argument-mismatch -ffixed-line-length-132 -ffast-math -funroll-loops -m64 -fopenmp < --- > FC = pgf90 > FFLAGS= -O -Mvect=sse -Mextend -Msecond_underscore -Bstatic_pgi -Mlfs 8,15c7,10 < < LIBS = -L/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/Linux2_x86_64 -lioapi \ < -L/opt/netcdf/netcdf-4/gnu/10.2.1/lib -lnetcdff -lnetcdf \ < -L/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib -fopenmp < < INCLUDE = -I/home/solange.luque/libraries/ioapi/v3.2-20200828/gnu/ioapi/fixed_src \ < -I/opt/netcdf/netcdf-4/gnu/10.2.1/include \ < -I/opt/openmpi/openmpi-4.1.2/gnu/10.2.1/lib/openmpi \ --- > LIBS = -L/usr/local/ioapi-3.2/Linux2_x86_64pg_gcc_nomp -lioapi \ > -L/usr/local/netcdf-4.7.0/lib -lnetcdff -lnetcdf > INCLUDE = -I/usr/local/src/ioapi-3.2/ioapi/fixed_src \ > -I//usr/local/netcdf-4.7.0/include \
Antes de compilar hay que hacerle unos cambios a algunos scripts.
/home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/src/MET2MGN diff readpar.f readpar.f.old 15,16c15 < INTEGER, parameter :: nlat=61 < INTEGER, parameter :: nlon=121 --- > parameter nlat=61,nlon=121 diff readmcip.f readmcip.f.old 38c38 < logical MCIP_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil,lfirst --- > logical MCIP_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil diff readwrfcamx.f readwrfcamx.f.old 37c37 < logical WRFCAMx_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil,lfirst --- > logical WRFCAMx_rad,SAT_par,ENVYN,INITIAL_HOUR,ifsoil
cd /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/src/IOAPI2UAM diff ioapi2uam.f ioapi2uam.f.old 165c165 < write(*,'(2(i9,f10.4))') ibdate,btime,iedate,etime --- > write(*,'(2(i,f))') ibdate,btime,iedate,etime 299c299 < write(*,'(a,2(i9,f10.4))')'Writing ',ibdate,btime,iedate,etime --- > write(*,'(a,2(i,f))')'Writing ',ibdate,btime,iedate,etime
Ahora sí compilamos.
cd ../.. source /opt/load-libs.sh 4 ./make_all_programs.scr &> make_gnu.log
Si no hay ningún error, deberían crearse los ejecutables en cada carpeta de cada programa.
[editar] Uso
Si bien MEGAN tiene varios programas (que son los que compilamos arriba), para usar con CMAQ el único que nos importa es TXT2IOAPI que agarra las salidas de los dos pre procesadores y las convierte de csv a netcdf (CT3.csv de prep4cmaq_cantype y OutputGridEF.csv del pre procesador de Python).
La idea es tener a la salida los siguientes archivo:
1) CT3.ncf: Este tiene la canopy fraction para seis tipos de vegetación (needleleaf trees, tropical forest trees, temperate broadleaf trees, shrubs, herbaceous plants, y crops)
2) LDF.ncf: Tiene "light dependence fractions" for 4 of the 19 MEGAN chemical species.
3) EFMAP.ncf: Factores de emisión para las 19 especies de MEGAN.
Con estos tres archivos, CMAQ ya se encarga de hacer todo.
El script para correr TXT2IOAPI está en:
cd /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work
Lo modificamos:
diff run.txt2ioapi.v32.csh run.txt2ioapi.v32.csh.old
6c6
< #foreach dom ( SE_US )
---
> foreach dom ( SE_US )
10,13c10,12
< #setenv EXEC $MGNEXE/$PROG
< setenv EXEC /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/src/TXT2IOAPI/txt2ioapi
< setenv GRIDDESC /home/solange.luque/estudios/CMAQ-AMBA/anidados_4_20ppb_20m/d01/spring/DATA/mcip/AMBA_11_d01/GRIDDESC
< setenv GDNAM3D ChemGBsAs_d01_cp
---
> setenv EXEC $MGNEXE/$PROG
> setenv GRIDDESC $MGNINP/MAP/GRIDDESC
> setenv GDNAM3D $dom
15c14
< #set dd = `echo $dom | cut -c 1-2`
---
> set dd = `echo $dom | cut -c 1-2`
20c19
< setenv EFSTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python/OutputGridEF.d01_spring.csv
---
> setenv EFSTXTF $MGNINP/MAP/OutputGridEF.$dom.csv
22c21
< setenv LDFTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN3_python/OutputGridEF.d01_spring.csv
---
> setenv LDFTXTF $MGNINP/MAP/OutputGridEF.$dom.csv
24c23
< setenv CTTXTF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGAN32_Prep_Code_Jan_2022/output/CT3.d01_spring.csv
---
> setenv CTTXTF $MGNINP/MAP/CT3.${GDNAM3D}.csv
28c27
< setenv LAIv_YN N
---
> setenv LAIv_YN Y
44,45c43,44
< setenv EFMAPS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/EFMAP.d01_spring.ncf
< setenv CANTYPFILE /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/CT3.d01_spring.ncf
---
> setenv EFMAPS $MGNINP/MAP/EFMAP.2019b.${GDNAM3D}.ncf
> setenv CANTYP $MGNINP/MAP/CT3_${GDNAM3D}.ncf
48c47
< setenv LDFILE /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/LDF.d01_spring.ncf
---
> setenv LDFILE $MGNINP/MAP/LDF_${GDNAM3D}.2019b.ncf
56c55
< setenv RUN_EFS T # [T|F]
---
> setenv RUN_EFS F # [T|F]
58c57
< setenv RUN_CANTYP T # [T|F]
---
> setenv RUN_CANTYP F # [T|F]
60c59
< setenv RUN_LDF T # [T|F]
---
> setenv RUN_LDF F # [T|F]
63c62
< setenv RUN_FERT F # [T|F] fertilizer data
---
> setenv RUN_FERT T # [T|F] fertilizer data
88c87
< #end
---
> end
Lo corremos:
qsub launch_txt2ioapi_gnu.pbs
Si todo sale bien esto debería generar en la carpeta output:
ls CT3.d01_spring.ncf EFMAP.d01_spring.ncf LDF.d01_spring.ncf
[editar] Acoplado con CMAQ
Las salidas de MEGAN para usar con CMAQ (CT3.ncf, EFMAP.ncf y LDF.ncf) son independientes del tiempo. Es decir, no importa para que período se las corra siempre son las mismas. Esto es porque es CMAQ después el encargado de considerar las condiciones meteorológicas y el LAI del período a considerar. Si no se usara CMAQ; sí se necesitarían usar los otros programas de MEGAN que no usamos que se encargarían de hacer esto mismo.
Lo único que hay que hacer parar correr CMAQ con MEGAN es cambiar la siguiente flag en el run_script.
setenv CTM_BIOGEMIS_MG Y
Y dar los paths de los archivos anteriores en:
setenv MEGAN_CTS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/CT3.d01_spring.ncf setenv MEGAN_EFS /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/EFMAP.d01_spring.ncf setenv MEGAN_LDF /home/solange.luque/MODELOS/Megan/MEGANv3.2_gnu/MEGANv3.21_Oct_2022/MEGANv3.2_Dec_2021/work/output/LDF.d01_spring.ncf
Si además queremos que CMAQ genere un fichero con las emisiones tenemos que modificar el CMAQ_Control_DESID.nml con lo siguiente:
cd /home/solange.luque/MODELOS/CMAQ/intel/src/CMAQ-master/CMAQ_Project/CCTM/scripts/BLD_CCTM_v54_intel cp CMAQ_Control_DESID.nml CMAQ_Control_DESID_megan.nml diff CMAQ_Control_DESID.nml CMAQ_Control_DESID_megan.nml 203c203 < Desid_N_Diag_Rules = 0 ! Exact Number of Diagnostic Rules Below --- > Desid_N_Diag_Rules = 1 ! Exact Number of Diagnostic Rules Below 239a240,248 > > ! Emisiones MEGAN > > Desid_Diag_Streams_Nml(1,:)= 'MIOG' > Desid_Diag_Fmt_Nml(1) = '3D' ! Options: LAYER1, COLSUM, 3D > Desid_Diag_Spec_Nml(1,:) = 'ALL' >
Esto va a generar un fichero CCTM_DESID1_MIOG* con las emisiones.
Y ahora en el run script (línea 333):
setenv DESID_CTRL_NML ${BLD}/CMAQ_Control_DESID_megan.nml